+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5uly | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of Human Protocadherin-15 EC2-3 | |||||||||
 Components Components | Protocadherin-15 | |||||||||
 Keywords Keywords | Calcium-binding protein / hearing / mechanotransduction / adhesion | |||||||||
| Function / homology |  Function and homology information Function and homology informationequilibrioception / sensory perception of light stimulus / stereocilium / photoreceptor cell maintenance / Sensory processing of sound by inner hair cells of the cochlea / Sensory processing of sound by outer hair cells of the cochlea / homophilic cell-cell adhesion / inner ear development / photoreceptor outer segment / sensory perception of sound ...equilibrioception / sensory perception of light stimulus / stereocilium / photoreceptor cell maintenance / Sensory processing of sound by inner hair cells of the cochlea / Sensory processing of sound by outer hair cells of the cochlea / homophilic cell-cell adhesion / inner ear development / photoreceptor outer segment / sensory perception of sound / cell adhesion / calcium ion binding / synapse / extracellular space / extracellular region / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.64 Å MOLECULAR REPLACEMENT / Resolution: 2.64 Å | |||||||||
 Authors Authors | Wimalasena, L.N. / Sotomayor, M. | |||||||||
| Funding support |  United States, 2items United States, 2items
| |||||||||
 Citation Citation |  Journal: Proc.Natl.Acad.Sci.USA / Year: 2020 Journal: Proc.Natl.Acad.Sci.USA / Year: 2020Title: Structural determinants of protocadherin-15 mechanics and function in hearing and balance perception. Authors: Choudhary, D. / Narui, Y. / Neel, B.L. / Wimalasena, L.N. / Klanseck, C.F. / De-la-Torre, P. / Chen, C. / Araya-Secchi, R. / Tamilselvan, E. / Sotomayor, M. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5uly.cif.gz 5uly.cif.gz | 371.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5uly.ent.gz pdb5uly.ent.gz | 304.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5uly.json.gz 5uly.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ul/5uly https://data.pdbj.org/pub/pdb/validation_reports/ul/5uly ftp://data.pdbj.org/pub/pdb/validation_reports/ul/5uly ftp://data.pdbj.org/pub/pdb/validation_reports/ul/5uly | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5tpkC  5w1dC  6bwnC  6bxuC  6e8fC  6eb5C  6eetC  6mfoC  6n22C  6n2eC  4apxS  5t4mS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 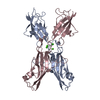
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments: Component-ID: _ / End auth comp-ID: ASP / End label comp-ID: ASP / Refine code: _
NCS ensembles :
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | Dimer as determined by SEC and AUC |
- Components
Components
| #1: Protein | Mass: 29375.723 Da / Num. of mol.: 4 / Fragment: residues 143-390 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PCDH15, USH1F / Production host: Homo sapiens (human) / Gene: PCDH15, USH1F / Production host:  #2: Chemical | ChemComp-CA / #3: Water | ChemComp-HOH / | Has protein modification | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.13 Å3/Da / Density % sol: 60.64 % |
|---|---|
| Crystal grow | Temperature: 277.15 K / Method: vapor diffusion, sitting drop / pH: 7.5 / Details: 0.1 M HEPES (pH 7.5), 0.1 M KCl, 15% PEG 6000 |
-Data collection
| Diffraction | Mean temperature: 100 K / Ambient temp details: oxford cryo-jet |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 24-ID-C / Wavelength: 0.9792 Å / Beamline: 24-ID-C / Wavelength: 0.9792 Å |
| Detector | Type: DECTRIS PILATUS 6M-F / Detector: PIXEL / Date: Mar 21, 2015 |
| Radiation | Monochromator: Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9792 Å / Relative weight: 1 |
| Reflection | Resolution: 2.64→50 Å / Num. obs: 42598 / % possible obs: 96.4 % / Redundancy: 2.6 % / Biso Wilson estimate: 45.7 Å2 / Rmerge(I) obs: 0.098 / Net I/σ(I): 10.625 |
| Reflection shell | Resolution: 2.64→2.69 Å / Redundancy: 2.4 % / Rmerge(I) obs: 0.581 / Mean I/σ(I) obs: 1.7 / % possible all: 95 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4APX, 5T4M Resolution: 2.64→50 Å / Cor.coef. Fo:Fc: 0.933 / Cor.coef. Fo:Fc free: 0.912 / SU B: 21.433 / SU ML: 0.215 / Cross valid method: THROUGHOUT / ESU R: 0.499 / ESU R Free: 0.288 / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 54.895 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 2.64→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj




