+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5l8x | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | X-RAY STRUCTURE OF APO METHANOCALDOCOCCUS JANNASCHII METHYLTRANSFERASE SUBUNIT A AT 1.85 ANGSTROM | ||||||
 要素 要素 | (Tetrahydromethanopterin S-methyltransferase subunit A) x 2 | ||||||
 キーワード キーワード | TRANSFERASE / Methanogenesis / Motor pump / Membrane protein / Methyltransferase / Cobalamin / Vitamin B12 / CoenzymeM / Rossmann fold / Hyperthermophile / Marine organism | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tetrahydromethanopterin S-methyltransferase / tetrahydromethanopterin S-methyltransferase activity / methanogenesis, from carbon dioxide / cobalt ion binding / one-carbon metabolic process / methylation / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |   Methanocaldococcus jannaschii (メタン生成菌) Methanocaldococcus jannaschii (メタン生成菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.85 Å 分子置換 / 解像度: 1.85 Å | ||||||
 データ登録者 データ登録者 | Wagner, T. / Ermler, U. / Shima, S. | ||||||
| 資金援助 |  日本, 1件 日本, 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2016 ジャーナル: Sci Rep / 年: 2016タイトル: MtrA of the sodium ion pumping methyltransferase binds cobalamin in a unique mode. 著者: Wagner, T. / Ermler, U. / Shima, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5l8x.cif.gz 5l8x.cif.gz | 144.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5l8x.ent.gz pdb5l8x.ent.gz | 112.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5l8x.json.gz 5l8x.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5l8x_validation.pdf.gz 5l8x_validation.pdf.gz | 461.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5l8x_full_validation.pdf.gz 5l8x_full_validation.pdf.gz | 462 KB | 表示 | |
| XML形式データ |  5l8x_validation.xml.gz 5l8x_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  5l8x_validation.cif.gz 5l8x_validation.cif.gz | 20.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l8/5l8x https://data.pdbj.org/pub/pdb/validation_reports/l8/5l8x ftp://data.pdbj.org/pub/pdb/validation_reports/l8/5l8x ftp://data.pdbj.org/pub/pdb/validation_reports/l8/5l8x | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18400.873 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: The HexaHistidine tagged in the N-terminus has been partially cleaved during limited proteolysis as the C-terminus. The original construct corresponds to MtrA 1-220. 由来: (組換発現)   Methanocaldococcus jannaschii (strain ATCC 43067 / DSM 2661 / JAL-1 / JCM 10045 / NBRC 100440) (メタン生成菌) Methanocaldococcus jannaschii (strain ATCC 43067 / DSM 2661 / JAL-1 / JCM 10045 / NBRC 100440) (メタン生成菌)遺伝子: mtrA, MJ0851 / プラスミド: pET28a 発現宿主:  参照: UniProt: Q58261, tetrahydromethanopterin S-methyltransferase |
|---|---|
| #2: タンパク質 | 分子量: 18457.926 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: The HexaHistidine tagged in the N-terminus has been partially cleaved during limited proteolysis as the C-terminus. The original construct corresponds to MtrA 1-220. 由来: (組換発現)   Methanocaldococcus jannaschii (strain ATCC 43067 / DSM 2661 / JAL-1 / JCM 10045 / NBRC 100440) (メタン生成菌) Methanocaldococcus jannaschii (strain ATCC 43067 / DSM 2661 / JAL-1 / JCM 10045 / NBRC 100440) (メタン生成菌)遺伝子: mtrA, MJ0851 / プラスミド: pET28a 発現宿主:  参照: UniProt: Q58261, tetrahydromethanopterin S-methyltransferase |
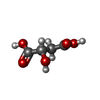
| #3: 化合物 | ChemComp-LMR / ( |
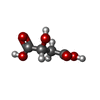
| #4: 化合物 | ChemComp-MLT / |
| #5: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.29 Å3/Da / 溶媒含有率: 46.42 % 解説: Tiny needles can have a maximum length of about 200 microns |
|---|---|
| 結晶化 | 温度: 281.15 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.6 詳細: 50 mg/ml protein after 2 months incubation, transparent needle-type crystals were reproducibly obtained in 2.2 M D/L-malate pH 7.6, 100 mM Tris-HCl pH 8.0 Temp details: Best crystals appeared at 281.15 K but smaller needles can appear at higher temperature too |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 1 Å / ビームライン: X10SA / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2013年10月14日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 1.85→45.11 Å / Num. obs: 25433 / % possible obs: 95.4 % / 冗長度: 2.3 % / CC1/2: 0.983 / Rmerge(I) obs: 0.123 / Rsym value: 0.093 / Net I/σ(I): 6.1 |
| 反射 シェル | 解像度: 1.85→1.95 Å / 冗長度: 2.3 % / Rmerge(I) obs: 0.609 / Mean I/σ(I) obs: 2 / % possible all: 95.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: MtrA cytoplasmic Methanothermus fervidus 解像度: 1.85→45.108 Å / SU ML: 0.23 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 23.92
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 24.6 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.85→45.108 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj