| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| Aimless | 0.5.2| データスケーリング | | PHASER | 2.5.7| 位相決定 | | PHENIX | 1.10.1| 精密化 | | PDB_EXTRACT | 3.2 | データ抽出 | | XDS | | データ削減 | | | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: 5KLF
解像度: 1.746→26.922 Å / SU ML: 0.18 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 21.24
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2073 | 412 | 5.4 % |
|---|
| Rwork | 0.1539 | - | - |
|---|
| obs | 0.1566 | 7635 | 99.17 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å |
|---|
| 原子変位パラメータ | Biso max: 48.25 Å2 / Biso mean: 17.0854 Å2 / Biso min: 8.32 Å2 |
|---|
| 精密化ステップ | サイクル: final / 解像度: 1.746→26.922 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 676 | 0 | 0 | 116 | 792 |
|---|
| Biso mean | - | - | - | 29.64 | - |
|---|
| 残基数 | - | - | - | - | 93 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.006 | 690 | | X-RAY DIFFRACTION | f_angle_d| 0.815 | 937 | | X-RAY DIFFRACTION | f_chiral_restr| 0.059 | 103 | | X-RAY DIFFRACTION | f_plane_restr| 0.004 | 121 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 7.846 | 393 | | | | | |
|
|---|
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 3 | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Num. reflection all | % reflection obs (%) |
|---|
| 1.7464-1.9991 | 0.2794 | 134 | 0.1859 | 2336 | 2470 | 98 | | 1.9991-2.5183 | 0.184 | 173 | 0.1479 | 2373 | 2546 | 100 | | 2.5183-26.9248 | 0.201 | 105 | 0.1478 | 2514 | 2619 | 100 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 1.4162 | -0.1035 | 0.3891 | 2.0687 | -0.7893 | 1.982 | -0.0315 | 0.163 | -0.0119 | 0.121 | 0.0308 | 0.0622 | -0.0697 | 0.0095 | 0.0048 | 0.1421 | -0.0172 | 0.0057 | 0.1415 | -0.0214 | 0.1044 | -5.3022 | -7.6245 | -20.3094 | | 2 | 2.6653 | -2.1064 | 1.7992 | 4.1887 | -0.1377 | 1.8642 | 0.0307 | 0.2852 | -0.2466 | -0.0876 | 0.0075 | 0.0776 | 0.1166 | 0.2012 | 0.0649 | 0.099 | 0.0236 | 0.0022 | 0.1418 | 0.0276 | 0.1352 | 2.0824 | -2.7958 | -18.3776 | | 3 | 2.7097 | 1.8009 | 0.0605 | 6.674 | -2.7144 | 4.5793 | 0.049 | 0.0388 | -0.0823 | -0.2533 | 0.0067 | 0.107 | 0.4681 | -0.0914 | -0.0641 | 0.1246 | -0.0151 | -0.0165 | 0.1085 | -0.0192 | 0.1381 | -7.6913 | -10.6979 | -16.083 | | 4 | 3.5288 | 0.1837 | 0.44 | 1.6429 | -0.7328 | 2.8911 | 0.106 | -0.1898 | -0.0691 | 0.0377 | -0.035 | 0.2117 | 0.3208 | -0.1464 | -0.075 | 0.1639 | -0.0287 | 0.0052 | 0.1176 | 0.015 | 0.1096 | -6.0013 | -10.5825 | -6.2028 | | 5 | 3.3225 | 0.0834 | 0.8127 | 4.3214 | -1.4117 | 2.8374 | 0.0344 | 0.199 | 0.0369 | 0.0044 | -0.2403 | -0.181 | -0.1381 | 0.1681 | 0.1711 | 0.1418 | -0.0345 | -0.0192 | 0.1446 | 0.02 | 0.1198 | 6.9348 | -1.3257 | -7.2491 | | 6 | 2.7853 | 0.1759 | 0.6035 | 2.5124 | -0.9451 | 2.3953 | -0.0357 | -0.062 | 0.128 | 0.1316 | 0.0085 | 0.0569 | -0.1555 | -0.1001 | 0.0627 | 0.1132 | 0.0017 | -0.0017 | 0.0996 | -0.0087 | 0.1055 | -5.3893 | -1.374 | -7.91 | | 7 | 4.6276 | 2.2498 | 1.0551 | 3.0932 | -0.425 | 1.8732 | -0.0501 | -0.1149 | 0.0757 | -0.0553 | 0.1152 | 0.2686 | 0.1233 | -0.12 | -0.089 | 0.0946 | 0.0183 | 0.0087 | 0.126 | -0.0094 | 0.1209 | -9.4688 | -5.0587 | -13.6178 | | 8 | 4.1379 | 4.9952 | -3.0206 | 8.3783 | -5.9175 | 5.2783 | -0.0523 | 0.1146 | -0.0838 | 0.0017 | 0.015 | -0.0151 | 0.1434 | 0.2197 | 0.0146 | 0.0929 | 0.0204 | -0.0003 | 0.1056 | 0.0123 | 0.1479 | 1.7227 | -6.6631 | -10.1613 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details | Auth asym-ID | Auth seq-ID |
|---|
| 1 | X-RAY DIFFRACTION | 1 | chain 'A' and (resid 335 through 352 )A| 335 - 352 | | 2 | X-RAY DIFFRACTION | 2 | chain 'A' and (resid 353 through 359 )A| 353 - 359 | | 3 | X-RAY DIFFRACTION | 3 | chain 'A' and (resid 360 through 365 )A| 360 - 365 | | 4 | X-RAY DIFFRACTION | 4 | chain 'A' and (resid 366 through 374 )A| 366 - 374 | | 5 | X-RAY DIFFRACTION | 5 | chain 'A' and (resid 375 through 379 )A| 375 - 379 | | 6 | X-RAY DIFFRACTION | 6 | chain 'A' and (resid 380 through 401 )A| 380 - 401 | | 7 | X-RAY DIFFRACTION | 7 | chain 'A' and (resid 402 through 416 )A| 402 - 416 | | 8 | X-RAY DIFFRACTION | 8 | chain 'A' and (resid 417 through 427 )A| 417 - 427 | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 uncultured bacterium (環境試料)
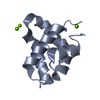
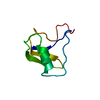
uncultured bacterium (環境試料) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.746 Å
分子置換 / 解像度: 1.746 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2016
ジャーナル: J.Biol.Chem. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5klc.cif.gz
5klc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5klc.ent.gz
pdb5klc.ent.gz PDB形式
PDB形式 5klc.json.gz
5klc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5klc_validation.pdf.gz
5klc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5klc_full_validation.pdf.gz
5klc_full_validation.pdf.gz 5klc_validation.xml.gz
5klc_validation.xml.gz 5klc_validation.cif.gz
5klc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kl/5klc
https://data.pdbj.org/pub/pdb/validation_reports/kl/5klc ftp://data.pdbj.org/pub/pdb/validation_reports/kl/5klc
ftp://data.pdbj.org/pub/pdb/validation_reports/kl/5klc リンク
リンク 集合体
集合体
 要素
要素 uncultured bacterium (環境試料) / 発現宿主:
uncultured bacterium (環境試料) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  LNLS
LNLS  / ビームライン: W01B-MX2 / 波長: 1.46 Å
/ ビームライン: W01B-MX2 / 波長: 1.46 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj
