| 登録情報 | データベース: PDB / ID: 5kbz
|
|---|
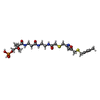
| タイトル | Structure of the PksA Product Template domain in complex with a phosphopantetheine mimetic |
|---|
 要素 要素 | Noranthrone synthase |
|---|
 キーワード キーワード | TRANSCRIPTION / Aflatoxin / Polyketide Synthase / Polyketide / Product Template / Dehydratase / Double Hot Dog Fold / Cyclase |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
noranthrone synthase / aflatoxin biosynthetic process / norsolorinate anthrone synthase activity / fatty acid synthase activity / phosphopantetheine binding / fatty acid biosynthetic process / identical protein binding類似検索 - 分子機能 Polyketide product template domain / Polyketide synthase dehydratase / Starter unit:ACP transacylase / Starter unit:ACP transacylase in aflatoxin biosynthesis / : / : / Polyketide and metazoan fatty acid synthase dehydratase (PKS/mFAS DH) domain profile. / Thioesterase / Thioesterase domain / Polyketide synthase dehydratase N-terminal domain ...Polyketide product template domain / Polyketide synthase dehydratase / Starter unit:ACP transacylase / Starter unit:ACP transacylase in aflatoxin biosynthesis / : / : / Polyketide and metazoan fatty acid synthase dehydratase (PKS/mFAS DH) domain profile. / Thioesterase / Thioesterase domain / Polyketide synthase dehydratase N-terminal domain / Polyketide synthase, dehydratase domain superfamily / Thiol Ester Dehydrase; Chain A / Malonyl-CoA ACP transacylase, ACP-binding / Acyl transferase domain superfamily / Acyl transferase / Acyl transferase domain / Acyl transferase domain in polyketide synthase (PKS) enzymes. / Acyl transferase/acyl hydrolase/lysophospholipase / Ketosynthase family 3 (KS3) domain profile. / Polyketide synthase, phosphopantetheine-binding domain / Phosphopantetheine attachment site / Beta-ketoacyl synthase / Polyketide synthase, beta-ketoacyl synthase domain / Beta-ketoacyl synthase, N-terminal / Beta-ketoacyl synthase, C-terminal / Beta-ketoacyl synthase, N-terminal domain / Beta-ketoacyl synthase, C-terminal domain / Thiolase-like / Phosphopantetheine attachment site / ACP-like superfamily / Carrier protein (CP) domain profile. / Phosphopantetheine binding ACP domain / Alpha/Beta hydrolase fold / Roll / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Aspergillus parasiticus (カビ) Aspergillus parasiticus (カビ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.803 Å 分子置換 / 解像度: 1.803 Å |
|---|
 データ登録者 データ登録者 | Tsai, S.C. / Burkart, M.D. / Townsend, C.A. / Barajas, J.F. / Shakya, G. / Topper, C.L. / Moreno, G. / Jackson, D.R. / Rivera, H. / La Clair, J.J. / Vagstad, A. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM 100738-02 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
タイトル: Polyketide mimetics yield structural and mechanistic insights into product template domain function in nonreducing polyketide synthases.
著者: Barajas, J.F. / Shakya, G. / Moreno, G. / Rivera, H. / Jackson, D.R. / Topper, C.L. / Vagstad, A.L. / La Clair, J.J. / Townsend, C.A. / Burkart, M.D. / Tsai, S.C. |
|---|
| 履歴 | | 登録 | 2016年6月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年5月10日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年5月24日 | Group: Database references |
|---|
| 改定 1.2 | 2017年6月7日 | Group: Database references |
|---|
| 改定 1.3 | 2017年9月27日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.5 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.803 Å
分子置換 / 解像度: 1.803 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5kbz.cif.gz
5kbz.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5kbz.ent.gz
pdb5kbz.ent.gz PDB形式
PDB形式 5kbz.json.gz
5kbz.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5kbz_validation.pdf.gz
5kbz_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5kbz_full_validation.pdf.gz
5kbz_full_validation.pdf.gz 5kbz_validation.xml.gz
5kbz_validation.xml.gz 5kbz_validation.cif.gz
5kbz_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kb/5kbz
https://data.pdbj.org/pub/pdb/validation_reports/kb/5kbz ftp://data.pdbj.org/pub/pdb/validation_reports/kb/5kbz
ftp://data.pdbj.org/pub/pdb/validation_reports/kb/5kbz
 リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.2.1 / 波長: 0.99997 Å
/ ビームライン: 8.2.1 / 波長: 0.99997 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj