[English] 日本語
 Yorodumi
Yorodumi- PDB-5jid: Crystal Structure of Human Transthyretin in Complex with Perfluor... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5jid | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of Human Transthyretin in Complex with Perfluorooctanoic acid (PFOA) | ||||||
 Components Components | Transthyretin | ||||||
 Keywords Keywords | TRANSPORT PROTEIN / thyroxine binding / Perfluorooctanoic acid(PFOA)complex | ||||||
| Function / homology |  Function and homology information Function and homology informationDefective visual phototransduction due to STRA6 loss of function / negative regulation of glomerular filtration / The canonical retinoid cycle in rods (twilight vision) / purine nucleobase metabolic process / hormone binding / Non-integrin membrane-ECM interactions / molecular sequestering activity / phototransduction, visible light / retinoid metabolic process / Retinoid metabolism and transport ...Defective visual phototransduction due to STRA6 loss of function / negative regulation of glomerular filtration / The canonical retinoid cycle in rods (twilight vision) / purine nucleobase metabolic process / hormone binding / Non-integrin membrane-ECM interactions / molecular sequestering activity / phototransduction, visible light / retinoid metabolic process / Retinoid metabolism and transport / hormone activity / azurophil granule lumen / Amyloid fiber formation / Neutrophil degranulation / protein-containing complex binding / protein-containing complex / extracellular space / extracellular exosome / extracellular region / identical protein binding Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.2 Å MOLECULAR REPLACEMENT / Resolution: 1.2 Å | ||||||
 Authors Authors | Begum, A. / Zhang, J. / Olofsson, A. / Andersson, P. / Sauer-Eriksson, A.E. | ||||||
| Funding support |  Sweden, 1items Sweden, 1items
| ||||||
 Citation Citation |  Journal: Environ. Sci. Technol. / Year: 2016 Journal: Environ. Sci. Technol. / Year: 2016Title: Structure-Based Virtual Screening Protocol for in Silico Identification of Potential Thyroid Disrupting Chemicals Targeting Transthyretin. Authors: Zhang, J. / Begum, A. / Brannstrom, K. / Grundstrom, C. / Iakovleva, I. / Olofsson, A. / Sauer-Eriksson, A.E. / Andersson, P.L. #1:  Journal: Plos One / Year: 2016 Journal: Plos One / Year: 2016Title: Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer Authors: Iakovleva, I. / Begum, A. / Brannstrom, K. / Wijesekera, A. / Nilsson, L. / Zhang, J. / Andersson, P. / Sauer-Eriksson, A.E. / Olofsson, A. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5jid.cif.gz 5jid.cif.gz | 157.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5jid.ent.gz pdb5jid.ent.gz | 127.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5jid.json.gz 5jid.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ji/5jid https://data.pdbj.org/pub/pdb/validation_reports/ji/5jid ftp://data.pdbj.org/pub/pdb/validation_reports/ji/5jid ftp://data.pdbj.org/pub/pdb/validation_reports/ji/5jid | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5jimC  5jiqC  5l4fC  5l4iC  5l4jC  5l4mC  1f41S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
| ||||||||
| Components on special symmetry positions |
|
- Components
Components
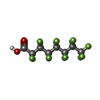
| #1: Protein | Mass: 13777.360 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: TTR, PALB / Production host: Homo sapiens (human) / Gene: TTR, PALB / Production host:  #2: Chemical | #3: Chemical | ChemComp-NA / | #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.1 Å3/Da / Density % sol: 41.5 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, hanging drop Details: Purified TTRwt was dialyzed against 10 mM sodiumphosphate buffer with 100 mM KCl pH 7.6 and concentrated to 5 mg per ml. PFOA was added at 5 x molar excess to the protein. The reservoir ...Details: Purified TTRwt was dialyzed against 10 mM sodiumphosphate buffer with 100 mM KCl pH 7.6 and concentrated to 5 mg per ml. PFOA was added at 5 x molar excess to the protein. The reservoir contained 1.3 to 1.6 M sodium citrate and 3.5 percent glycerol at pH 5.5. Drop size 3 plus 3 microliter. PH range: 7.6 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID29 / Wavelength: 0.913 Å / Beamline: ID29 / Wavelength: 0.913 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Feb 24, 2015 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.913 Å / Relative weight: 1 |
| Reflection | Resolution: 1.2→40 Å / Num. obs: 71054 / % possible obs: 96.6 % / Redundancy: 8.6 % / Rmerge(I) obs: 0.048 / Net I/σ(I): 19.26 |
| Reflection shell | Resolution: 1.2→1.24 Å / Redundancy: 6.1 % / Rmerge(I) obs: 0.787 / Mean I/σ(I) obs: 1.79 / % possible all: 75.14 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 1F41 Resolution: 1.2→38.183 Å / SU ML: 0.1 / Cross valid method: FREE R-VALUE / σ(F): 1.35 / Phase error: 15.22
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.2→38.183 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller




















 PDBj
PDBj