+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5h9v | ||||||
|---|---|---|---|---|---|---|---|
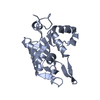
| タイトル | Crystal structure of Regnase PIN domain, form I | ||||||
 要素 要素 | Ribonuclease ZC3H12A | ||||||
 キーワード キーワード | HYDROLASE / RNase | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報immune response-activating signaling pathway / positive regulation of miRNA catabolic process / positive regulation of protein deubiquitination / negative regulation of muscle cell apoptotic process / RNA exonuclease activity / miRNA catabolic process / rough endoplasmic reticulum membrane / negative regulation of T-helper 17 cell differentiation / positive regulation of mRNA catabolic process / host-mediated suppression of viral genome replication ...immune response-activating signaling pathway / positive regulation of miRNA catabolic process / positive regulation of protein deubiquitination / negative regulation of muscle cell apoptotic process / RNA exonuclease activity / miRNA catabolic process / rough endoplasmic reticulum membrane / negative regulation of T-helper 17 cell differentiation / positive regulation of mRNA catabolic process / host-mediated suppression of viral genome replication / cellular response to sodium arsenite / negative regulation of cardiac muscle contraction / cellular response to ionomycin / 3'-UTR-mediated mRNA destabilization / nuclease activity / negative regulation of nitric oxide biosynthetic process / positive regulation of lipid storage / cellular response to chemokine / mRNA 3'-UTR AU-rich region binding / positive regulation of p38MAPK cascade / negative regulation of non-canonical NF-kappaB signal transduction / miRNA binding / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / negative regulation of interleukin-1 beta production / mRNA catabolic process / negative regulation of protein phosphorylation / protein complex oligomerization / negative regulation of interleukin-6 production / negative regulation of type II interferon production / negative regulation of tumor necrosis factor production / cellular response to interleukin-1 / positive regulation of execution phase of apoptosis / protein deubiquitination / positive regulation of fat cell differentiation / cellular response to glucose starvation / rough endoplasmic reticulum / positive regulation of defense response to virus by host / negative regulation of canonical NF-kappaB signal transduction / positive regulation of autophagy / negative regulation of cytokine production involved in inflammatory response / RNA endonuclease activity / positive regulation of endothelial cell migration / mRNA 3'-UTR binding / P-body / cellular response to virus / positive regulation of protein import into nucleus / RNA stem-loop binding / positive regulation of reactive oxygen species metabolic process / positive regulation of angiogenesis / cellular response to tumor necrosis factor / nervous system development / T cell receptor signaling pathway / ribosome binding / cellular response to lipopolysaccharide / cellular response to oxidative stress / angiogenesis / 加水分解酵素; エステル加水分解酵素 / cell differentiation / cysteine-type deubiquitinase activity / cytoskeleton / inflammatory response / negative regulation of gene expression / mRNA binding / apoptotic process / DNA damage response / chromatin binding / positive regulation of gene expression / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.75 Å 多波長異常分散 / 解像度: 2.75 Å | ||||||
 データ登録者 データ登録者 | Yokogawa, M. / Tsushima, T. / Adachi, W. / Noda, N.N. / Inagaki, F. | ||||||
| 資金援助 |  日本, 1件 日本, 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2016 ジャーナル: Sci Rep / 年: 2016タイトル: Structural basis for the regulation of enzymatic activity of Regnase-1 by domain-domain interactions 著者: Yokogawa, M. / Tsushima, T. / Noda, N.N. / Kumeta, H. / Enokizono, Y. / Yamashita, K. / Standley, D.M. / Takeuchi, O. / Akira, S. / Inagaki, F. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5h9v.cif.gz 5h9v.cif.gz | 284.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5h9v.ent.gz pdb5h9v.ent.gz | 233.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5h9v.json.gz 5h9v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5h9v_validation.pdf.gz 5h9v_validation.pdf.gz | 463.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5h9v_full_validation.pdf.gz 5h9v_full_validation.pdf.gz | 474.2 KB | 表示 | |
| XML形式データ |  5h9v_validation.xml.gz 5h9v_validation.xml.gz | 27.1 KB | 表示 | |
| CIF形式データ |  5h9v_validation.cif.gz 5h9v_validation.cif.gz | 34.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h9/5h9v https://data.pdbj.org/pub/pdb/validation_reports/h9/5h9v ftp://data.pdbj.org/pub/pdb/validation_reports/h9/5h9v ftp://data.pdbj.org/pub/pdb/validation_reports/h9/5h9v | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 24717.869 Da / 分子数: 4 / 断片: PIN domain, UNP residues 134-339 / 由来タイプ: 組換発現 / 由来: (組換発現)   参照: UniProt: Q5D1E7, 加水分解酵素; エステル加水分解酵素 #2: 化合物 | ChemComp-NA / Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.52 Å3/Da / 溶媒含有率: 65.03 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 / 詳細: 1 M (NH4)2HPO4, 200 mM NaCl, 100 mM sodium citrate |
-データ収集
| 回折 | 平均測定温度: 95 K | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Photon Factory Photon Factory  / ビームライン: AR-NE3A / 波長: 0.97899, 0.97928, 0.96405 / ビームライン: AR-NE3A / 波長: 0.97899, 0.97928, 0.96405 | ||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 270 / 検出器: CCD / 日付: 2009年4月8日 | ||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||
| 放射波長 |
| ||||||||||||
| 反射 | 解像度: 2.75→47.49 Å / Num. obs: 33467 / % possible obs: 90.5 % / 冗長度: 7.9 % / Net I/σ(I): 22.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2.75→47.49 Å / Cor.coef. Fo:Fc: 0.954 / Cor.coef. Fo:Fc free: 0.932 / SU B: 25.849 / SU ML: 0.22 / 交差検証法: THROUGHOUT / ESU R: 0.386 / ESU R Free: 0.267 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS 多波長異常分散 / 解像度: 2.75→47.49 Å / Cor.coef. Fo:Fc: 0.954 / Cor.coef. Fo:Fc free: 0.932 / SU B: 25.849 / SU ML: 0.22 / 交差検証法: THROUGHOUT / ESU R: 0.386 / ESU R Free: 0.267 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 92.716 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.75→47.49 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj
