| 登録情報 | データベース: PDB / ID: 5cok
|
|---|
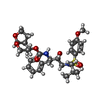
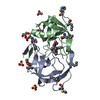
| タイトル | X-ray crystal structure of wild type HIV-1 protease in complex with GRL-0476 |
|---|
 要素 要素 | HIV-1 protease |
|---|
 キーワード キーワード | Hydrolase/Hydrolase Inhibitor / GRL-0476 / HIV-1 protease / protease-inhibitor / darunavir / Tp-THF / nonpeptidic / Hydrolase-Hydrolase Inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
aspartic-type endopeptidase activity / proteolysis類似検索 - 分子機能 Retropepsin-like catalytic domain / Retropepsins / Retroviral aspartyl protease / Aspartyl protease, retroviral-type family profile. / Peptidase A2A, retrovirus, catalytic / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site / Eukaryotic and viral aspartyl proteases active site. / Aspartic peptidase domain superfamily ...Retropepsin-like catalytic domain / Retropepsins / Retroviral aspartyl protease / Aspartyl protease, retroviral-type family profile. / Peptidase A2A, retrovirus, catalytic / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site / Eukaryotic and viral aspartyl proteases active site. / Aspartic peptidase domain superfamily / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 (3aS,4S,7aR)-hexahydro-4H-furo[2,3-b]pyran-4-yl / Pol protein / Pol protein類似検索 - 構成要素 |
|---|
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.801 Å 分子置換 / 解像度: 1.801 Å |
|---|
 データ登録者 データ登録者 | Yedidi, R.S. / Hayashi, H. / Aoki, M. / Das, D. / Ghosh, A.K. / Mitsuya, H. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: J.Virol. / 年: 2015 ジャーナル: J.Virol. / 年: 2015
タイトル: C-5-Modified Tetrahydropyrano-Tetrahydofuran-Derived Protease Inhibitors (PIs) Exert Potent Inhibition of the Replication of HIV-1 Variants Highly Resistant to Various PIs, including Darunavir.
著者: Aoki, M. / Hayashi, H. / Yedidi, R.S. / Martyr, C.D. / Takamatsu, Y. / Aoki-Ogata, H. / Nakamura, T. / Nakata, H. / Das, D. / Yamagata, Y. / Ghosh, A.K. / Mitsuya, H. |
|---|
| 履歴 | | 登録 | 2015年7月20日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年1月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年2月24日 | Group: Database references |
|---|
| 改定 1.2 | 2017年9月27日 | Group: Author supporting evidence / Data collection ...Author supporting evidence / Data collection / Database references / Derived calculations
カテゴリ: citation / diffrn_source ...citation / diffrn_source / pdbx_audit_support / pdbx_struct_oper_list
Item: _citation.journal_id_CSD / _diffrn_source.pdbx_synchrotron_site ..._citation.journal_id_CSD / _diffrn_source.pdbx_synchrotron_site / _pdbx_audit_support.funding_organization / _pdbx_struct_oper_list.symmetry_operation |
|---|
| 改定 1.3 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2024年3月6日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.801 Å
分子置換 / 解像度: 1.801 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: J.Virol. / 年: 2015
ジャーナル: J.Virol. / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5cok.cif.gz
5cok.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5cok.ent.gz
pdb5cok.ent.gz PDB形式
PDB形式 5cok.json.gz
5cok.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5cok_validation.pdf.gz
5cok_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5cok_full_validation.pdf.gz
5cok_full_validation.pdf.gz 5cok_validation.xml.gz
5cok_validation.xml.gz 5cok_validation.cif.gz
5cok_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/co/5cok
https://data.pdbj.org/pub/pdb/validation_reports/co/5cok ftp://data.pdbj.org/pub/pdb/validation_reports/co/5cok
ftp://data.pdbj.org/pub/pdb/validation_reports/co/5cok リンク
リンク 集合体
集合体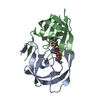
 要素
要素
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL44XU / 波長: 0.9 Å
/ ビームライン: BL44XU / 波長: 0.9 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 1.801→45.282 Å / FOM work R set: 0.8133 / SU ML: 0.19 / 交差検証法: FREE R-VALUE / σ(F): 0.64 / 位相誤差: 25.71 / 立体化学のターゲット値: ML
分子置換 / 解像度: 1.801→45.282 Å / FOM work R set: 0.8133 / SU ML: 0.19 / 交差検証法: FREE R-VALUE / σ(F): 0.64 / 位相誤差: 25.71 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj