[English] 日本語
 Yorodumi
Yorodumi- PDB-5cbl: Crystal structure of the C-terminal domain of human galectin-4 wi... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5cbl | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of the C-terminal domain of human galectin-4 with lactose | ||||||||||||
 Components Components | (Galectin-4) x 2 | ||||||||||||
 Keywords Keywords | SUGAR BINDING PROTEIN / galectins / beta-galactosides binding | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationantibacterial peptide biosynthetic process / galactoside binding / : / carbohydrate binding / cell adhesion / extracellular space / plasma membrane / cytosol Similarity search - Function | ||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.781 Å molecular replacement / Resolution: 1.781 Å | ||||||||||||
 Authors Authors | Rustiguel, J.K. / Nonato, M.C. | ||||||||||||
| Funding support |  Brazil, 1items Brazil, 1items
| ||||||||||||
 Citation Citation |  Journal: Protein Expr.Purif. / Year: 2015 Journal: Protein Expr.Purif. / Year: 2015Title: Recombinant expression, purification and preliminary biophysical and structural studies of C-terminal carbohydrate recognition domain from human galectin-4. Authors: Rustiguel, J.K. / Kumagai, P.S. / Dias-Baruffi, M. / Costa-Filho, A.J. / Nonato, M.C. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5cbl.cif.gz 5cbl.cif.gz | 218.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5cbl.ent.gz pdb5cbl.ent.gz | 174.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5cbl.json.gz 5cbl.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/cb/5cbl https://data.pdbj.org/pub/pdb/validation_reports/cb/5cbl ftp://data.pdbj.org/pub/pdb/validation_reports/cb/5cbl ftp://data.pdbj.org/pub/pdb/validation_reports/cb/5cbl | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  3ojbS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 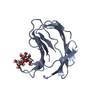
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments: Component-ID: 1 / Ens-ID: 1 / End auth comp-ID: ILE / End label comp-ID: ILE
|
- Components
Components
-Protein , 2 types, 4 molecules ACDB
| #1: Protein | Mass: 16375.621 Da / Num. of mol.: 3 / Fragment: UNP residues 179-323 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: LGALS4 / Plasmid: pET-28a-SUMO / Production host: Homo sapiens (human) / Gene: LGALS4 / Plasmid: pET-28a-SUMO / Production host:  #2: Protein | | Mass: 16451.740 Da / Num. of mol.: 1 / Fragment: UNP residues 179-323 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: LGALS4 / Plasmid: pET-28a-SUMO / Production host: Homo sapiens (human) / Gene: LGALS4 / Plasmid: pET-28a-SUMO / Production host:  |
|---|
-Sugars , 1 types, 4 molecules
| #3: Polysaccharide | beta-D-galactopyranose-(1-4)-beta-D-glucopyranose / beta-lactose |
|---|
-Non-polymers , 3 types, 300 molecules 




| #4: Chemical | ChemComp-SO4 / | ||
|---|---|---|---|
| #5: Chemical | | #6: Water | ChemComp-HOH / | |
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.98 Å3/Da / Density % sol: 37.99 % / Description: twinning crystals |
|---|---|
| Crystal grow | Temperature: 294 K / Method: vapor diffusion, sitting drop / pH: 8.5 Details: 0.1 M Tris-HCl pH 8.5, 25% a 35% PEG 4000-10000, 0.2 to 0.4 M lithium or ammonium sulfate PH range: 8.5 |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRL SSRL  / Beamline: BL12-2 / Wavelength: 0.9795 Å / Beamline: BL12-2 / Wavelength: 0.9795 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: May 18, 2015 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9795 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Redundancy: 5.9 % / Number: 274222 / Rsym value: 0.101 / D res high: 1.781 Å / D res low: 63.37 Å / Num. obs: 46238 / % possible obs: 95.4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection twin | Operator: l,-k,h / Fraction: 0.27 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.781→63.37 Å / Num. obs: 46238 / % possible obs: 95.4 % / Redundancy: 5.9 % / Rpim(I) all: 0.044 / Rrim(I) all: 0.111 / Rsym value: 0.101 / Net I/av σ(I): 4.5 / Net I/σ(I): 9.1 / Num. measured all: 274222 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
-Phasing
| Phasing | Method:  molecular replacement molecular replacement | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR | Model details: Phaser MODE: MR_AUTO
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3OJB Resolution: 1.781→63.37 Å / Cross valid method: FREE R-VALUE / σ(F): 1.36 / Phase error: 29.7 / Stereochemistry target values: TWIN_LSQ_F
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 101.21 Å2 / Biso mean: 29.8378 Å2 / Biso min: 9.8 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.781→63.37 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 14
|
 Movie
Movie Controller
Controller












 PDBj
PDBj






