登録情報 データベース : PDB / ID : 4x1pタイトル The crystal structure of mupain-1-17 in complex with murinised human uPA at pH4.6 MUPAIN-1-17 Urokinase-type plasminogen activator キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)synthetic construct (人工物) 手法 / / 解像度 : 1.6 Å データ登録者 Jiang, L. / Zhao, B. / Xu, P. / Andreasen, P. / Huang, M. 資金援助 組織 認可番号 国 National Natural Science Foundation of China 31161130356 National Natural Science Foundation of China 31170707 the Natural Science Foundation of Fujian Province 2012J05071 the Danish National Research Foundation 26-331-6
ジャーナル : Int.J.Biochem.Cell Biol. / 年 : 2015タイトル : Distinctive binding modes and inhibitory mechanisms of two peptidic inhibitors of urokinase-type plasminogen activator with isomeric P1 residues.著者 : Jiang, L. / Zhao, B. / Xu, P. / Srensen, H.P. / Jensen, J.K. / Christensen, A. / Hosseini, M. / Nielsen, N.C. / Jensen, K.J. / Andreasen, P.A. / Huang, M. 履歴 登録 2014年11月25日 登録サイト / 処理サイト 改定 1.0 2015年10月21日 Provider / タイプ 改定 1.1 2017年10月18日 Group / Derived calculations / カテゴリ / pdbx_struct_oper_listItem / _pdbx_struct_oper_list.symmetry_operation改定 1.2 2025年4月9日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Structure summary カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id 改定 1.3 2025年9月17日 Group / Derived calculations / Structure summaryカテゴリ database_PDB_caveat / pdbx_modification_feature ... database_PDB_caveat / pdbx_modification_feature / pdbx_validate_close_contact / struct_conn
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.6 Å
シンクロトロン / 解像度: 1.6 Å  データ登録者
データ登録者 中国,
中国,  デンマーク, 4件
デンマーク, 4件  引用
引用 ジャーナル: Int.J.Biochem.Cell Biol. / 年: 2015
ジャーナル: Int.J.Biochem.Cell Biol. / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4x1p.cif.gz
4x1p.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4x1p.ent.gz
pdb4x1p.ent.gz PDB形式
PDB形式 4x1p.json.gz
4x1p.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/x1/4x1p
https://data.pdbj.org/pub/pdb/validation_reports/x1/4x1p ftp://data.pdbj.org/pub/pdb/validation_reports/x1/4x1p
ftp://data.pdbj.org/pub/pdb/validation_reports/x1/4x1p リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: PLAU / プラスミド: pPICZaA / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PLAU / プラスミド: pPICZaA / 発現宿主:  Pichia pastoris (菌類) / 株 (発現宿主): X33 / 参照: UniProt: P00749, u-plasminogen activator
Pichia pastoris (菌類) / 株 (発現宿主): X33 / 参照: UniProt: P00749, u-plasminogen activator

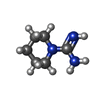




 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 0.979 Å
/ ビームライン: BL17U / 波長: 0.979 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー











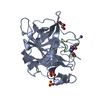
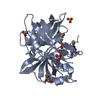





 PDBj
PDBj



