+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4ufb | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the Angiotensin-1 converting enzyme N-domain in complex with Lys-Pro | |||||||||
 要素 要素 | ANGIOTENSIN-CONVERTING ENZYME | |||||||||
 キーワード キーワード | HYDROLASE / ANGIOTENSIN-CONVERTING ENZYME / METALLOPROTEASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mononuclear cell proliferation / cell proliferation in bone marrow / bradykinin receptor binding / exopeptidase activity / regulation of angiotensin metabolic process / substance P catabolic process / tripeptidyl-peptidase activity / peptidyl-dipeptidase A / regulation of renal output by angiotensin / negative regulation of gap junction assembly ...mononuclear cell proliferation / cell proliferation in bone marrow / bradykinin receptor binding / exopeptidase activity / regulation of angiotensin metabolic process / substance P catabolic process / tripeptidyl-peptidase activity / peptidyl-dipeptidase A / regulation of renal output by angiotensin / negative regulation of gap junction assembly / hormone catabolic process / bradykinin catabolic process / metallodipeptidase activity / regulation of smooth muscle cell migration / regulation of hematopoietic stem cell proliferation / neutrophil mediated immunity / hormone metabolic process / mitogen-activated protein kinase binding / mitogen-activated protein kinase kinase binding / chloride ion binding / arachidonate secretion / post-transcriptional regulation of gene expression / peptide catabolic process / heart contraction / antigen processing and presentation of peptide antigen via MHC class I / positive regulation of systemic arterial blood pressure / regulation of heart rate by cardiac conduction / regulation of systemic arterial blood pressure by renin-angiotensin / blood vessel remodeling / amyloid-beta metabolic process / hematopoietic stem cell differentiation / peptidyl-dipeptidase activity / regulation of vasoconstriction / Metabolism of Angiotensinogen to Angiotensins / angiotensin maturation / metallocarboxypeptidase activity / blood vessel diameter maintenance / angiotensin-activated signaling pathway / kidney development / regulation of synaptic plasticity / metalloendopeptidase activity / regulation of blood pressure / male gonad development / metallopeptidase activity / peptidase activity / actin binding / spermatogenesis / endopeptidase activity / calmodulin binding / lysosome / endosome / negative regulation of gene expression / external side of plasma membrane / proteolysis / extracellular space / extracellular exosome / extracellular region / zinc ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å | |||||||||
 データ登録者 データ登録者 | Masuyer, G. / Douglas, R.G. / Sturrock, E.D. / Acharya, K.R. | |||||||||
 引用 引用 |  ジャーナル: Sci.Rep. / 年: 2015 ジャーナル: Sci.Rep. / 年: 2015タイトル: Structural Basis of Ac-Sdkp Hydrolysis by Angiotensin-I Converting Enzyme 著者: Masuyer, G. / Douglas, R.G. / Sturrock, E.D. / Acharya, K.R. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4ufb.cif.gz 4ufb.cif.gz | 1008.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4ufb.ent.gz pdb4ufb.ent.gz | 843.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4ufb.json.gz 4ufb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4ufb_validation.pdf.gz 4ufb_validation.pdf.gz | 5.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4ufb_full_validation.pdf.gz 4ufb_full_validation.pdf.gz | 5.3 MB | 表示 | |
| XML形式データ |  4ufb_validation.xml.gz 4ufb_validation.xml.gz | 101 KB | 表示 | |
| CIF形式データ |  4ufb_validation.cif.gz 4ufb_validation.cif.gz | 147 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uf/4ufb https://data.pdbj.org/pub/pdb/validation_reports/uf/4ufb ftp://data.pdbj.org/pub/pdb/validation_reports/uf/4ufb ftp://data.pdbj.org/pub/pdb/validation_reports/uf/4ufb | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 72606.508 Da / 分子数: 4 / 断片: N DOMAIN (UNP RESIDUES 30-657) / 変異: YES / 由来タイプ: 組換発現 / 詳細: MINIMALLY GLYCOSYLATED MUTANT / 由来: (組換発現)  HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): CHO K1 HOMO SAPIENS (ヒト) / 細胞株 (発現宿主): CHO K1発現宿主:  参照: UniProt: P12821, peptidyl-dipeptidase A |
|---|
-糖 , 4種, 12分子
| #2: 多糖 | alpha-L-fucopyranose-(1-6)-2-acetamido-2-deoxy-beta-D-glucopyranose #3: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose #4: 多糖 | #5: 多糖 | |
|---|
-非ポリマー , 7種, 1643分子 
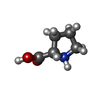











| #6: 化合物 | ChemComp-LYS / #7: 化合物 | ChemComp-PRO / #8: 化合物 | ChemComp-ZN / #9: 化合物 | ChemComp-CL / #10: 化合物 | ChemComp-PEG / #11: 化合物 | ChemComp-P6G / #12: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|---|
| 配列の詳細 | P62328 RESIDUES 4-5 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3 Å3/Da / 溶媒含有率: 58 % / 解説: NONE |
|---|---|
| 結晶化 | pH: 8.5 詳細: 0.06 M DIVALENT CATIONS, 0.1 M TRIS/BICINE PH 8.5, 30 % PEG550MME/PEG20000 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 0.9763 / ビームライン: I03 / 波長: 0.9763 |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2014年11月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9763 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→30.3 Å / Num. obs: 293868 / % possible obs: 97.2 % / Observed criterion σ(I): 0 / 冗長度: 3.2 % / Rmerge(I) obs: 0.1 / Net I/σ(I): 6.2 |
| 反射 シェル | 解像度: 1.8→1.9 Å / 冗長度: 3.2 % / Rmerge(I) obs: 0.7 / Mean I/σ(I) obs: 1.5 / % possible all: 96 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 3NXQ 解像度: 1.8→113.94 Å / Cor.coef. Fo:Fc: 0.961 / Cor.coef. Fo:Fc free: 0.947 / SU B: 8.043 / SU ML: 0.117 / 交差検証法: THROUGHOUT / ESU R: 0.13 / ESU R Free: 0.122 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. DI-PEPTIDE LYS-PRO IS PRODUCT OF AC-SDKP CLEAVAGE REACTION BY ACE
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 29.206 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→113.94 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj








