+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4uea | ||||||
|---|---|---|---|---|---|---|---|
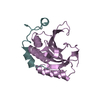
| タイトル | Complex of D. melanogaster eIF4E with a designed 4E-binding protein (Form I) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSLATION / GENE REGULATION / CAP BINDING PROTEIN / 4E BINDING PROTEIN / TRANSLATIONAL REPRESSION | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TOR signaling pathway / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / ISG15 antiviral mechanism / Transport of the SLBP independent Mature mRNA / Transport of the SLBP Dependant Mature mRNA / Transport of Mature mRNA Derived from an Intronless Transcript / L13a-mediated translational silencing of Ceruloplasmin expression / mTORC1-mediated signalling / Translation initiation complex formation / Ribosomal scanning and start codon recognition ...TOR signaling pathway / Activation of the mRNA upon binding of the cap-binding complex and eIFs, and subsequent binding to 43S / ISG15 antiviral mechanism / Transport of the SLBP independent Mature mRNA / Transport of the SLBP Dependant Mature mRNA / Transport of Mature mRNA Derived from an Intronless Transcript / L13a-mediated translational silencing of Ceruloplasmin expression / mTORC1-mediated signalling / Translation initiation complex formation / Ribosomal scanning and start codon recognition / muscle cell postsynaptic specialization / RNA metabolic process / neuronal ribonucleoprotein granule / eukaryotic initiation factor 4G binding / RNA cap binding / eukaryotic translation initiation factor 4F complex / RNA 7-methylguanosine cap binding / translation initiation factor activity / translational initiation / neuromuscular junction / P-body / regulation of translation / nuclear body / translation / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  SYNTHETIC CONSTRUCT (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.62 Å 分子置換 / 解像度: 2.62 Å | ||||||
 データ登録者 データ登録者 | Peter, D. / Weichenrieder, O. | ||||||
 引用 引用 |  ジャーナル: Mol.Cell / 年: 2015 ジャーナル: Mol.Cell / 年: 2015タイトル: Molecular Architecture of 4E-BP Translational Inhibitors Bound to Eif4E. 著者: Peter, D. / Igreja, C. / Weber, R. / Wohlbold, L. / Weiler, C. / Ebertsch, L. / Weichenrieder, O. / Izaurralde, E. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4uea.cif.gz 4uea.cif.gz | 237.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4uea.ent.gz pdb4uea.ent.gz | 196.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4uea.json.gz 4uea.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4uea_validation.pdf.gz 4uea_validation.pdf.gz | 454.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4uea_full_validation.pdf.gz 4uea_full_validation.pdf.gz | 455.9 KB | 表示 | |
| XML形式データ |  4uea_validation.xml.gz 4uea_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  4uea_validation.cif.gz 4uea_validation.cif.gz | 28.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ue/4uea https://data.pdbj.org/pub/pdb/validation_reports/ue/4uea ftp://data.pdbj.org/pub/pdb/validation_reports/ue/4uea ftp://data.pdbj.org/pub/pdb/validation_reports/ue/4uea | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 21249.037 Da / 分子数: 3 / 断片: UNP RESIDUES 80-259 / 由来タイプ: 組換発現 由来: (組換発現)  プラスミド: PETMCN (PNEA) / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 5103.927 Da / 分子数: 3 / 断片: RESIDUES 1-36 / 由来タイプ: 組換発現 / 由来: (組換発現) SYNTHETIC CONSTRUCT (人工物) / プラスミド: PETMCN (PNEA) / 発現宿主:  配列の詳細 | THE FIRST FOUR RESIDUES OF CHAIN A, C, E REMAIN FROM THE EXPRESSION TAG. COMPARED TO UNP P48598, ...THE FIRST FOUR RESIDUES OF CHAIN A, C, E REMAIN FROM THE EXPRESSION | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 48 % / 解説: NONE |
|---|---|
| 結晶化 | pH: 8.5 詳細: 0.03M EACH DIETHYLENE GLYCOL, TRIETHYLENE GLYCOL, TETRAETHYLENE GLYCOL, PENTAETHYLENE GLYCOL, 0.1M BICINE/TRIZMA BASE PH 8.5, 10% PEG8000, 20% ETHYLENE GLYCOL |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 1 / ビームライン: X10SA / 波長: 1 |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2014年10月6日 / 詳細: DYNAMICALLY BENDABLE MIRRORS |
| 放射 | モノクロメーター: SI(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.62→47.3 Å / Num. obs: 20025 / % possible obs: 99.6 % / Observed criterion σ(I): -3 / 冗長度: 4.9 % / Biso Wilson estimate: 63.49 Å2 / Rsym value: 0.07 / Net I/σ(I): 14.4 |
| 反射 シェル | 解像度: 2.62→2.69 Å / 冗長度: 5 % / Mean I/σ(I) obs: 2.1 / Rsym value: 0.85 / % possible all: 99.5 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 4UE8 CHAIN A 解像度: 2.62→47.27 Å / SU ML: 0.4 / σ(F): 1.35 / 位相誤差: 31.3 / 立体化学のターゲット値: ML 詳細: HYDROGENS WERE REFINED IN THE RIDING POSITIONS. THE SIDECHAINS OF THE FOLLOWING RESIDUES WERE TRUNCATED AT C-BETA ATOMS. CHAIN A, RESIDUES 68, 218, 219, 220, 222, 223, 224, 236. CHAIN B, ...詳細: HYDROGENS WERE REFINED IN THE RIDING POSITIONS. THE SIDECHAINS OF THE FOLLOWING RESIDUES WERE TRUNCATED AT C-BETA ATOMS. CHAIN A, RESIDUES 68, 218, 219, 220, 222, 223, 224, 236. CHAIN B, RESIDUE 0. CHAIN C, RESIDUES 152, 190, 227. CHAIN E, RESIDUES 222, 223. THE FOLLOWING RESIDUES ARE DISORDERED. CHAIN A, RESIDUES 237 TO 248. CHAIN C, RESIDUES 236 TO 248. CHAIN E, RESIDUES 235 TO 248.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 70.1 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.62→47.27 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj
