[English] 日本語
 Yorodumi
Yorodumi- PDB-4tju: Crystal Structure of human Tankyrase 2 in complex with 3,4-CPQ-5-C. -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4tju | ||||||
|---|---|---|---|---|---|---|---|
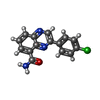
| Title | Crystal Structure of human Tankyrase 2 in complex with 3,4-CPQ-5-C. | ||||||
 Components Components | Tankyrase-2 | ||||||
 Keywords Keywords | TRANSFERASE / poly(ADP-ribosylation) polymerase (PARP) | ||||||
| Function / homology |  Function and homology information Function and homology informationXAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity / pericentriolar material / Transferases; Glycosyltransferases; Pentosyltransferases / NAD+ poly-ADP-ribosyltransferase activity / positive regulation of telomere maintenance via telomerase / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / Wnt signaling pathway / Regulation of PTEN stability and activity / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / nuclear envelope / chromosome, telomeric region / Ub-specific processing proteases / Golgi membrane / perinuclear region of cytoplasm / enzyme binding / metal ion binding / nucleus / cytoplasm / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / Resolution: 1.57 Å SYNCHROTRON / Resolution: 1.57 Å | ||||||
 Authors Authors | Qiu, W. / Lam, R. / Romanov, V. / Gordon, R. / Gebremeskel, S. / Vodsedalek, J. / Thompson, C. / Beletskaya, I. / Battaile, K.P. / Pai, E.F. / Chirgadze, N.Y. | ||||||
 Citation Citation |  Journal: Acta Crystallogr.,Sect.D / Year: 2014 Journal: Acta Crystallogr.,Sect.D / Year: 2014Title: Insights into the binding of PARP inhibitors to the catalytic domain of human tankyrase-2. Authors: Qiu, W. / Lam, R. / Voytyuk, O. / Romanov, V. / Gordon, R. / Gebremeskel, S. / Vodsedalek, J. / Thompson, C. / Beletskaya, I. / Battaile, K.P. / Pai, E.F. / Rottapel, R. / Chirgadze, N.Y. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4tju.cif.gz 4tju.cif.gz | 200.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4tju.ent.gz pdb4tju.ent.gz | 156.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4tju.json.gz 4tju.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/tj/4tju https://data.pdbj.org/pub/pdb/validation_reports/tj/4tju ftp://data.pdbj.org/pub/pdb/validation_reports/tj/4tju ftp://data.pdbj.org/pub/pdb/validation_reports/tj/4tju | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4pmlC  4pnlC  4pnmC  4pnnC  4pnqC  4pnrC  4pnsC  4pntC  4tjwC  4tjyC  4tk0C  4tk5C  4tkfC  4tkgC  4tkiC C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 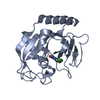
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| Unit cell |
| ||||||||
| Details | 1 |
- Components
Components
| #1: Protein | Mass: 25986.291 Da / Num. of mol.: 4 / Fragment: PARP, catalytic domain Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host: Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host:  #2: Chemical | ChemComp-ZN / #3: Chemical | ChemComp-CNQ / #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.17 Å3/Da / Density % sol: 43.42 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 7.5 Details: 0.2M NaCl, 0.1M Sodium, Hepes buffer at pH7.5, 12-15% iso-propanol |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 17-ID / Wavelength: 1 Å / Beamline: 17-ID / Wavelength: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: ADSC QUANTUM 210 / Detector: CCD / Date: Aug 12, 2009 / Details: mirrors | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: GRAPHITE / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.57→79.49 Å / Num. obs: 117003 / % possible obs: 92.1 % / Redundancy: 6.16 % / Biso Wilson estimate: 20.35 Å2 / Rmerge(I) obs: 0.056 / Net I/σ(I): 19.91 / Num. measured all: 794005 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1 / Rejects: _
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 1.57→76.68 Å / Cor.coef. Fo:Fc: 0.9489 / Cor.coef. Fo:Fc free: 0.9318 / SU R Cruickshank DPI: 0.091 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.098 / SU Rfree Blow DPI: 0.094 / SU Rfree Cruickshank DPI: 0.09
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 112 Å2 / Biso mean: 26.35 Å2 / Biso min: 9.55 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.187 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.57→76.68 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.57→1.61 Å / Total num. of bins used: 20
|
 Movie
Movie Controller
Controller











 PDBj
PDBj