+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4rcm | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
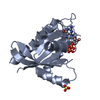
| タイトル | Crystal structure of the Pho92 YTH domain in complex with m6A | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | RNA BINDING PROTEIN/RNA / Structural Genomics / Structural Genomics Consortium / SGC / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of phosphate metabolic process / N6-methyladenosine-containing RNA reader activity / mRNA destabilization / regulation of mRNA stability / mRNA 3'-UTR binding / mRNA binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  synthetic construct (人工物) | |||||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å | |||||||||
 データ登録者 データ登録者 | Tempel, W. / Xu, C. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / Min, J. / Structural Genomics Consortium (SGC) | |||||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2015 ジャーナル: J.Biol.Chem. / 年: 2015タイトル: Structural Basis for the Discriminative Recognition of N6-Methyladenosine RNA by the Human YT521-B Homology Domain Family of Proteins. 著者: Xu, C. / Liu, K. / Ahmed, H. / Loppnau, P. / Schapira, M. / Min, J. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4rcm.cif.gz 4rcm.cif.gz | 84.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4rcm.ent.gz pdb4rcm.ent.gz | 61.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4rcm.json.gz 4rcm.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4rcm_validation.pdf.gz 4rcm_validation.pdf.gz | 462.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4rcm_full_validation.pdf.gz 4rcm_full_validation.pdf.gz | 464.5 KB | 表示 | |
| XML形式データ |  4rcm_validation.xml.gz 4rcm_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  4rcm_validation.cif.gz 4rcm_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rc/4rcm https://data.pdbj.org/pub/pdb/validation_reports/rc/4rcm ftp://data.pdbj.org/pub/pdb/validation_reports/rc/4rcm ftp://data.pdbj.org/pub/pdb/validation_reports/rc/4rcm | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | AS PER THE AUTHORS THE BIOLOGICAL ASSEMBLY IS UNKNOWN |
- 要素
要素
| #1: タンパク質 | 分子量: 19313.057 Da / 分子数: 2 / 断片: UNP residues 141-306 / 由来タイプ: 組換発現 由来: (組換発現)  株: ATCC 204508 / S288c / 遺伝子: PHO92, MRB1, YDR374C / プラスミド: pET28-MHL / 発現宿主:  #2: RNA鎖 | 分子量: 1560.995 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) #3: 化合物 | ChemComp-UNX / #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.84 Å3/Da / 溶媒含有率: 33.29 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 詳細: 20% PEG-3350, 0.2 M potassium isocyanate, vapor diffusion, sitting drop, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU FR-E DW / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU FR-E DW / 波長: 1.5418 Å | ||||||||||||||||||
| 検出器 | タイプ: RIGAKU RAXIS / 検出器: IMAGE PLATE / 日付: 2014年4月15日 | ||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 | ||||||||||||||||||
| 反射 | 解像度: 1.8→38.4 Å / Num. obs: 27985 / % possible obs: 99.5 % / 冗長度: 3.6 % / Rmerge(I) obs: 0.09 / Net I/σ(I): 10.9 | ||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / 冗長度: 3.3 %
|
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRIES 4R3H AND 4RCI 解像度: 1.8→32.97 Å / Cor.coef. Fo:Fc: 0.937 / Cor.coef. Fo:Fc free: 0.895 / WRfactor Rfree: 0.2485 / WRfactor Rwork: 0.1999 / Occupancy max: 1 / Occupancy min: 0.3 / FOM work R set: 0.747 / SU B: 4.912 / SU ML: 0.143 / SU R Cruickshank DPI: 0.1674 / SU Rfree: 0.1578 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.167 / ESU R Free: 0.158 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: AN ENSEMBLE OF CRYSTALLOGRAPHIC MODELS OF HUMAN YTHDC1 AND YTHDF1 WAS USED AS SEARCH MODEL FOR MOLECULAR REPLACEMENT. PARROT WAS USED FOR DENSITY MODIFICATION. ARP/WARP WAS USED FOR PHASE ...詳細: AN ENSEMBLE OF CRYSTALLOGRAPHIC MODELS OF HUMAN YTHDC1 AND YTHDF1 WAS USED AS SEARCH MODEL FOR MOLECULAR REPLACEMENT. PARROT WAS USED FOR DENSITY MODIFICATION. ARP/WARP WAS USED FOR PHASE IMPROVEMENT AND AUTOMATED MODEL BUILDING. COOT WAS USED FOR INTERACTIVE MODEL BUILDING. MODEL GEOMETRY WAS EVALUATED WITH MOLPROBITY.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 66.6 Å2 / Biso mean: 19.2124 Å2 / Biso min: 2 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→32.97 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.8→1.85 Å / Total num. of bins used: 20 /
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj


