+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4pd4 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural analysis of atovaquone-inhibited cytochrome bc1 complex reveals the molecular basis of antimalarial drug action | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | OXIDOREDUCTASE/INHIBITOR / Cytochrome bc1 complex / Membrane protein complex / antimalarial drug / inhibitor / OXIDOREDUCTASE-INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Complex III assembly / : / matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / Respiratory electron transport / Mitochondrial protein degradation / mitochondrial respiratory chain complex III assembly / cellular respiration / respiratory chain complex III / quinol-cytochrome-c reductase ...Complex III assembly / : / matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / Respiratory electron transport / Mitochondrial protein degradation / mitochondrial respiratory chain complex III assembly / cellular respiration / respiratory chain complex III / quinol-cytochrome-c reductase / quinol-cytochrome-c reductase activity / immunoglobulin complex / mitochondrial electron transport, ubiquinol to cytochrome c / mitochondrial crista / proton transmembrane transport / nuclear periphery / aerobic respiration / metalloendopeptidase activity / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / adaptive immune response / oxidoreductase activity / mitochondrial inner membrane / immune response / heme binding / mitochondrion / proteolysis / extracellular region / metal ion binding / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.04 Å 分子置換 / 解像度: 3.04 Å | |||||||||
 データ登録者 データ登録者 | Birth, D. / Kao, W.-C. / Hunte, C. | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2014 ジャーナル: Nat Commun / 年: 2014タイトル: Structural analysis of atovaquone-inhibited cytochrome bc1 complex reveals the molecular basis of antimalarial drug action. 著者: Birth, D. / Kao, W.C. / Hunte, C. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4pd4.cif.gz 4pd4.cif.gz | 466.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4pd4.ent.gz pdb4pd4.ent.gz | 363.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4pd4.json.gz 4pd4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4pd4_validation.pdf.gz 4pd4_validation.pdf.gz | 2.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4pd4_full_validation.pdf.gz 4pd4_full_validation.pdf.gz | 2.9 MB | 表示 | |
| XML形式データ |  4pd4_validation.xml.gz 4pd4_validation.xml.gz | 88.2 KB | 表示 | |
| CIF形式データ |  4pd4_validation.cif.gz 4pd4_validation.cif.gz | 116.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pd/4pd4 https://data.pdbj.org/pub/pdb/validation_reports/pd/4pd4 ftp://data.pdbj.org/pub/pdb/validation_reports/pd/4pd4 ftp://data.pdbj.org/pub/pdb/validation_reports/pd/4pd4 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-Cytochrome b-c1 complex subunit ... , 7種, 7分子 ABEFGHI
| #1: タンパク質 | 分子量: 47445.242 Da / 分子数: 1 / 断片: UNP residues 27-457 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P07256 |
|---|---|
| #2: タンパク質 | 分子量: 38751.918 Da / 分子数: 1 / 断片: UNP residues 17-368 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P07257 |
| #5: タンパク質 | 分子量: 20122.955 Da / 分子数: 1 / 断片: UNP residues 31-215 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P08067, quinol-cytochrome-c reductase |
| #6: タンパク質 | 分子量: 8854.792 Da / 分子数: 1 / 断片: UNP residues 74-147 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00127 |
| #7: タンパク質 | 分子量: 14452.557 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00128 |
| #8: タンパク質 | 分子量: 10856.314 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P08525 |
| #9: タンパク質 | 分子量: 6535.484 Da / 分子数: 1 / 断片: UNP residues 2-58 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P22289 |
-タンパク質 , 2種, 2分子 CD
| #3: タンパク質 | 分子量: 43686.590 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P00163 |
|---|---|
| #4: タンパク質 | 分子量: 27807.395 Da / 分子数: 1 / 断片: UNP residues 62-309 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P07143 |
-抗体 , 2種, 2分子 JK
| #10: 抗体 | 分子量: 14365.817 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #11: 抗体 | 分子量: 11926.350 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
-非ポリマー , 7種, 11分子 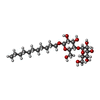


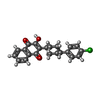









| #12: 化合物 | ChemComp-UMQ / | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| #13: 化合物 | | #14: 化合物 | #15: 化合物 | ChemComp-AOQ / | #16: 化合物 | ChemComp-3PE / | #17: 化合物 | ChemComp-UQ6 / | #18: 化合物 | ChemComp-FES / | |
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.24 Å3/Da / 溶媒含有率: 70.96 % |
|---|---|
| 結晶化 | 温度: 277.2 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.8 詳細: polyethylene glycol 4000, DMSO, Sucrose, Potassium phosphate, n-undecyl-?-D-maltopyranoside, atovaquone |
-データ収集
| 回折 | 平均測定温度: 105 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06DA / 波長: 1 Å / ビームライン: X06DA / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS PILATUS 2M-F / 検出器: PIXEL / 日付: 2012年6月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 3.04→25 Å / Num. obs: 77221 / % possible obs: 98.2 % / 冗長度: 6.8 % / Biso Wilson estimate: 97.24 Å2 / Net I/σ(I): 15.1 |
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR |
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 3.04→24.989 Å / SU ML: 0.52 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 39.2 / 立体化学のターゲット値: ML 分子置換 / 解像度: 3.04→24.989 Å / SU ML: 0.52 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 39.2 / 立体化学のターゲット値: ML詳細: Author states that the structure was determined by molecular replacement and used the high-resolution structure of yeast cyt bc1 complex (pdb 3cx5) as basis for refinement. Quality of x-ray ...詳細: Author states that the structure was determined by molecular replacement and used the high-resolution structure of yeast cyt bc1 complex (pdb 3cx5) as basis for refinement. Quality of x-ray diffraction data for the new structure is limited and some poorly resolved loops were set to zero occupancy. The large complex covers a wide range of B-factors, with the highest in the matrix core subunits (chains A and B) and the lowest in the membrane integral subunit cytochrome b (chain C). In former structures obtained at room-temperature , such as 2KB9 at 2.3 angstrom, B factors range from above140 for chain A to below 30 for chain C. In this structure, the overall B-factors are lower but cover a similar range with very low up to zero B factors in the best ordered regions.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 130.91 Å2 / Biso mean: 45.1046 Å2 / Biso min: 0 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 3.04→24.989 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 14
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj



















