| 登録情報 | データベース: PDB / ID: 4ozc
|
|---|
| タイトル | Backbone Modifications in the Protein GB1 Helix and Loops: beta-ACPC21, beta-ACPC24, beta-3-Lys28, beta-3-Lys31, beta-ACPC35, beta-ACPC40 |
|---|
 要素 要素 | Streptococcal Protein GB1 Backbone Modified Variant: beta-ACPC21, beta-ACPC24, beta-3-Lys28, beta-3-Lys31, beta-ACPC35, beta-ACPC40 |
|---|
 キーワード キーワード | DE NOVO PROTEIN / unnatural backbone |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
IgG-binding B / B domain / Ubiquitin-like (UB roll) - #10 / M protein-type anchor domain / GA-like domain / GA-like domain / Immunoglobulin/albumin-binding domain superfamily / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. ...IgG-binding B / B domain / Ubiquitin-like (UB roll) - #10 / M protein-type anchor domain / GA-like domain / GA-like domain / Immunoglobulin/albumin-binding domain superfamily / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain / Ubiquitin-like (UB roll) / Roll / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Streptococcus sp. (バクテリア) Streptococcus sp. (バクテリア) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.301 Å 分子置換 / 解像度: 2.301 Å |
|---|
 データ登録者 データ登録者 | Reinert, Z.E. / Horne, W.S. |
|---|
 引用 引用 |  ジャーナル: Chem Sci / 年: 2014 ジャーナル: Chem Sci / 年: 2014
タイトル: Folding Thermodynamics of Protein-Like Oligomers with Heterogeneous Backbones.
著者: Reinert, Z.E. / Horne, W.S. |
|---|
| 履歴 | | 登録 | 2014年2月14日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年7月16日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年9月24日 | Group: Database references |
|---|
| 改定 1.2 | 2014年12月24日 | Group: Database references |
|---|
| 改定 1.3 | 2017年11月22日 | Group: Derived calculations / Refinement description / Source and taxonomy
カテゴリ: pdbx_entity_src_syn / pdbx_struct_oper_list / software
Item: _pdbx_entity_src_syn.pdbx_alt_source_flag / _pdbx_struct_oper_list.symmetry_operation |
|---|
| 改定 1.4 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 2.0 | 2023年11月15日 | Group: Atomic model / Data collection
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / pdbx_validate_main_chain_plane / pdbx_validate_peptide_omega / pdbx_validate_torsion
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp_atom.atom_id / _chem_comp_bond.atom_id_1 / _chem_comp_bond.atom_id_2 / _pdbx_validate_peptide_omega.auth_comp_id_1 / _pdbx_validate_peptide_omega.auth_comp_id_2 / _pdbx_validate_peptide_omega.auth_seq_id_1 / _pdbx_validate_peptide_omega.auth_seq_id_2 / _pdbx_validate_peptide_omega.omega |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Streptococcus sp. (バクテリア)
Streptococcus sp. (バクテリア) X線回折 /
X線回折 /  分子置換 / 解像度: 2.301 Å
分子置換 / 解像度: 2.301 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chem Sci / 年: 2014
ジャーナル: Chem Sci / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4ozc.cif.gz
4ozc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4ozc.ent.gz
pdb4ozc.ent.gz PDB形式
PDB形式 4ozc.json.gz
4ozc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4ozc_validation.pdf.gz
4ozc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4ozc_full_validation.pdf.gz
4ozc_full_validation.pdf.gz 4ozc_validation.xml.gz
4ozc_validation.xml.gz 4ozc_validation.cif.gz
4ozc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/oz/4ozc
https://data.pdbj.org/pub/pdb/validation_reports/oz/4ozc ftp://data.pdbj.org/pub/pdb/validation_reports/oz/4ozc
ftp://data.pdbj.org/pub/pdb/validation_reports/oz/4ozc リンク
リンク 集合体
集合体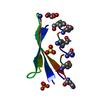
 要素
要素 Streptococcus sp. (バクテリア) / 参照: UniProt: P19909*PLUS
Streptococcus sp. (バクテリア) / 参照: UniProt: P19909*PLUS X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





