| 登録情報 | データベース: PDB / ID: 4od9
|
|---|
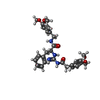
| タイトル | Structure of Cathepsin D with inhibitor N-(3,4-dimethoxybenzyl)-Nalpha-{N-[(3,4-dimethoxyphenyl)acetyl]carbamimidoyl}-D-phenylalaninamide |
|---|
 要素 要素 | - Cathepsin D heavy chain
- Cathepsin D light chain
|
|---|
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / LYSOSOMAL ASPARTIC PROTEASE / HYDROLASE-HYDROLASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cathepsin D / aspartic-type peptidase activity / insulin catabolic process / regulation of establishment of protein localization / lipoprotein catabolic process / execution phase of apoptosis / insulin receptor recycling / Collagen degradation / autophagosome assembly / Metabolism of Angiotensinogen to Angiotensins ...cathepsin D / aspartic-type peptidase activity / insulin catabolic process / regulation of establishment of protein localization / lipoprotein catabolic process / execution phase of apoptosis / insulin receptor recycling / Collagen degradation / autophagosome assembly / Metabolism of Angiotensinogen to Angiotensins / Insulin receptor recycling / MHC class II antigen presentation / lysosomal lumen / endosome lumen / antigen processing and presentation of exogenous peptide antigen via MHC class II / specific granule lumen / tertiary granule lumen / melanosome / peptidase activity / : / Estrogen-dependent gene expression / ficolin-1-rich granule lumen / aspartic-type endopeptidase activity / lysosome / endosome membrane / positive regulation of apoptotic process / membrane raft / lysosomal membrane / cysteine-type endopeptidase activity / Neutrophil degranulation / proteolysis / extracellular space / extracellular exosome / extracellular region / cytosol類似検索 - 分子機能 Cathepsin D / Aspartic peptidase, N-terminal / A1 Propeptide / Eukaryotic aspartyl protease / Aspartic peptidase A1 family / Peptidase family A1 domain / Peptidase family A1 domain profile. / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site ...Cathepsin D / Aspartic peptidase, N-terminal / A1 Propeptide / Eukaryotic aspartyl protease / Aspartic peptidase A1 family / Peptidase family A1 domain / Peptidase family A1 domain profile. / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site / Eukaryotic and viral aspartyl proteases active site. / Aspartic peptidase domain superfamily / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Graedler, U. / Czodrowski, P. / Tsaklakidis, C. / Klein, M. / Maskos, K. / Leuthner, B. |
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2014 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2014
タイトル: Structure-based optimization of non-peptidic Cathepsin D inhibitors.
著者: Gradler, U. / Czodrowski, P. / Tsaklakidis, C. / Klein, M. / Werkmann, D. / Lindemann, S. / Maskos, K. / Leuthner, B. |
|---|
| 履歴 | | 登録 | 2014年1月10日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年8月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年9月10日 | Group: Database references |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.id / _atom_site_anisotrop.pdbx_auth_asym_id / _atom_site_anisotrop.pdbx_auth_seq_id / _atom_site_anisotrop.pdbx_label_asym_id / _chem_comp.name / _chem_comp.type / _pdbx_entity_nonpoly.comp_id / _pdbx_entity_nonpoly.entity_id / _pdbx_entity_nonpoly.name / _pdbx_struct_assembly_gen.asym_id_list / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2024年11月27日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2014
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4od9.cif.gz
4od9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4od9.ent.gz
pdb4od9.ent.gz PDB形式
PDB形式 4od9.json.gz
4od9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4od9_validation.pdf.gz
4od9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4od9_full_validation.pdf.gz
4od9_full_validation.pdf.gz 4od9_validation.xml.gz
4od9_validation.xml.gz 4od9_validation.cif.gz
4od9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/od/4od9
https://data.pdbj.org/pub/pdb/validation_reports/od/4od9 ftp://data.pdbj.org/pub/pdb/validation_reports/od/4od9
ftp://data.pdbj.org/pub/pdb/validation_reports/od/4od9 リンク
リンク 集合体
集合体

 要素
要素 Homo sapiens (ヒト) / 遺伝子: CPSD, CTSD
Homo sapiens (ヒト) / 遺伝子: CPSD, CTSD
 Homo sapiens (ヒト) / 遺伝子: CPSD, CTSD
Homo sapiens (ヒト) / 遺伝子: CPSD, CTSD





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 0.999 Å
/ ビームライン: X06DA / 波長: 0.999 Å 解析
解析 分子置換 / 解像度: 1.9→38 Å / Cor.coef. Fo:Fc: 0.9239 / Cor.coef. Fo:Fc free: 0.9104 / SU R Cruickshank DPI: 0.143 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
分子置換 / 解像度: 1.9→38 Å / Cor.coef. Fo:Fc: 0.9239 / Cor.coef. Fo:Fc free: 0.9104 / SU R Cruickshank DPI: 0.143 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj


















