| 登録情報 | データベース: PDB / ID: 4o95
|
|---|
| タイトル | Crystal structure of maize cytokinin oxidase/dehydrogenase 4 (ZmCKO4) in complex with phenylurea inhibitor CPPU |
|---|
 要素 要素 | Cytokinin dehydrogenase 4 |
|---|
 キーワード キーワード | OXIDOREDUCTASE / CYTOKININ / FLAVOPROTEIN / PHENYL-UREA INHIBITOR |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cytokinin dehydrogenase / cytokinin dehydrogenase activity / cytokinin metabolic process / FAD binding類似検索 - 分子機能 Cytokinin dehydrogenase 1, FAD/cytokinin binding domain / Cytokinin dehydrogenase 1, FAD and cytokinin binding / : / FAD-linked oxidases, C-terminal domain / Oxygen oxidoreductase covalent FAD-binding site / Oxygen oxidoreductases covalent FAD-binding site. / Cytokinin dehydrogenase, C-terminal domain superfamily / Vanillyl-alcohol Oxidase; Chain A, domain 3 / FAD-linked oxidase-like, C-terminal / Uridine Diphospho-n-acetylenolpyruvylglucosamine Reductase, domain 2 ...Cytokinin dehydrogenase 1, FAD/cytokinin binding domain / Cytokinin dehydrogenase 1, FAD and cytokinin binding / : / FAD-linked oxidases, C-terminal domain / Oxygen oxidoreductase covalent FAD-binding site / Oxygen oxidoreductases covalent FAD-binding site. / Cytokinin dehydrogenase, C-terminal domain superfamily / Vanillyl-alcohol Oxidase; Chain A, domain 3 / FAD-linked oxidase-like, C-terminal / Uridine Diphospho-n-acetylenolpyruvylglucosamine Reductase, domain 2 / Uridine Diphospho-n-acetylenolpyruvylglucosamine Reductase; domain 2 / Uridine Diphospho-n-acetylenolpyruvylglucosamine Reductase; domain 3 - #10 / Uridine Diphospho-n-acetylenolpyruvylglucosamine Reductase; domain 3 / FAD linked oxidase, N-terminal / FAD binding domain / FAD-binding, type PCMH, subdomain 1 / FAD-binding domain, PCMH-type / PCMH-type FAD-binding domain profile. / FAD-binding, type PCMH, subdomain 2 / FAD-binding, type PCMH-like superfamily / 2-Layer Sandwich / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 1-(2-chloropyridin-4-yl)-3-phenylurea / FLAVIN-ADENINE DINUCLEOTIDE / DI(HYDROXYETHYL)ETHER / cytokinin dehydrogenase類似検索 - 構成要素 |
|---|
| 生物種 |   Zea mays (トウモロコシ) Zea mays (トウモロコシ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.75 Å 分子置換 / 解像度: 1.75 Å |
|---|
 データ登録者 データ登録者 | Kopecny, D. / Morera, S. / Vigouroux, A. / Koncitikova, R. |
|---|
 引用 引用 |  ジャーナル: Febs J. / 年: 2016 ジャーナル: Febs J. / 年: 2016
タイトル: Kinetic and structural investigation of the cytokinin oxidase/dehydrogenase active site.
著者: Kopecny, D. / Koncitikova, R. / Popelka, H. / Briozzo, P. / Vigouroux, A. / Kopecna, M. / Zalabak, D. / Sebela, M. / Skopalova, J. / Frebort, I. / Morera, S. |
|---|
| 履歴 | | 登録 | 2014年1月1日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2015年4月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年11月4日 | Group: Database references |
|---|
| 改定 1.2 | 2015年11月25日 | Group: Database references |
|---|
| 改定 1.3 | 2022年8月24日 | Group: Database references / Derived calculations
カテゴリ: citation / citation_author ...citation / citation_author / database_2 / struct_conn / struct_site
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation.year / _citation_author.identifier_ORCID / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2024年11月20日 | Group: Data collection / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.75 Å
分子置換 / 解像度: 1.75 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Febs J. / 年: 2016
ジャーナル: Febs J. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4o95.cif.gz
4o95.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4o95.ent.gz
pdb4o95.ent.gz PDB形式
PDB形式 4o95.json.gz
4o95.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4o95_validation.pdf.gz
4o95_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4o95_full_validation.pdf.gz
4o95_full_validation.pdf.gz 4o95_validation.xml.gz
4o95_validation.xml.gz 4o95_validation.cif.gz
4o95_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o9/4o95
https://data.pdbj.org/pub/pdb/validation_reports/o9/4o95 ftp://data.pdbj.org/pub/pdb/validation_reports/o9/4o95
ftp://data.pdbj.org/pub/pdb/validation_reports/o9/4o95 リンク
リンク 集合体
集合体
 要素
要素


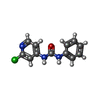





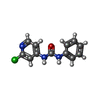





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.97934 Å
/ ビームライン: PROXIMA 1 / 波長: 0.97934 Å 解析
解析 分子置換 / 解像度: 1.75→42.67 Å / Cor.coef. Fo:Fc: 0.9519 / Cor.coef. Fo:Fc free: 0.9403 / SU R Cruickshank DPI: 0.099 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
分子置換 / 解像度: 1.75→42.67 Å / Cor.coef. Fo:Fc: 0.9519 / Cor.coef. Fo:Fc free: 0.9403 / SU R Cruickshank DPI: 0.099 / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj






