| 登録情報 | データベース: PDB / ID: 4hyf
|
|---|
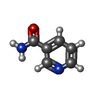
| タイトル | Structural basis and SAR for OD 270, a lead stage 1,2,4-triazole based specific Tankyrase1/2 inhibitor |
|---|
 要素 要素 | Tankyrase-2 |
|---|
 キーワード キーワード | TRANSFERASE/TRANSFERASE INHIBITOR / CATALYTIC FRAGMENT / PARP / ADP-RIBOSYLATION / ANK REPEAT / CHROMOSOMAL PROTEIN / GLYCOSYLTRANSFERASE / PHOSPHORYLATION / TELOMERE / TRANSFERASE / TRANSLOCATION / TRANSPORT / WNT-SIGNALLING / TRANSFERASE-TRANSFERASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity / pericentriolar material / NAD+ poly-ADP-ribosyltransferase activity / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / positive regulation of telomere maintenance via telomerase / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / Regulation of PTEN stability and activity / Wnt signaling pathway / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / nuclear envelope / chromosome, telomeric region / Ub-specific processing proteases / Golgi membrane / perinuclear region of cytoplasm / enzyme binding / metal ion binding / nucleus / cytosol / cytoplasm類似検索 - 分子機能 Ankyrin repeat / Phosphoenolpyruvate Carboxykinase; domain 3 - #10 / Phosphoenolpyruvate Carboxykinase; domain 3 / Poly(ADP-ribose) polymerase catalytic domain / Poly(ADP-ribose) polymerase, catalytic domain / PARP catalytic domain profile. / SAM domain (Sterile alpha motif) / SAM domain profile. / Sterile alpha motif. / Sterile alpha motif domain ...Ankyrin repeat / Phosphoenolpyruvate Carboxykinase; domain 3 - #10 / Phosphoenolpyruvate Carboxykinase; domain 3 / Poly(ADP-ribose) polymerase catalytic domain / Poly(ADP-ribose) polymerase, catalytic domain / PARP catalytic domain profile. / SAM domain (Sterile alpha motif) / SAM domain profile. / Sterile alpha motif. / Sterile alpha motif domain / Sterile alpha motif/pointed domain superfamily / Ankyrin repeat / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / Alpha-Beta Complex / Alpha Beta類似検索 - ドメイン・相同性 Chem-1AK / NICOTINAMIDE / Poly [ADP-ribose] polymerase tankyrase-2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å |
|---|
 データ登録者 データ登録者 | Perdreau, H. / Ekblad, B. / Voronkov, A. / Holsworth, D.D. / Waaler, J. / Drewes, G. / Schueler, H. / Krauss, S. / Morth, J.P. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2013 ジャーナル: J.Med.Chem. / 年: 2013
タイトル: Structural Basis and SAR for G007-LK, a Lead Stage 1,2,4-Triazole Based Specific Tankyrase 1/2 Inhibitor.
著者: Voronkov, A. / Holsworth, D.D. / Waaler, J. / Wilson, S.R. / Ekblad, B. / Perdreau-Dahl, H. / Dinh, H. / Drewes, G. / Hopf, C. / Morth, J.P. / Krauss, S. |
|---|
| 履歴 | | 登録 | 2012年11月13日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2013年3月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年5月1日 | Group: Database references |
|---|
| 改定 1.2 | 2017年11月15日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 1.3 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_label_asym_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.8 Å
分子置換 / 解像度: 2.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2013
ジャーナル: J.Med.Chem. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4hyf.cif.gz
4hyf.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4hyf.ent.gz
pdb4hyf.ent.gz PDB形式
PDB形式 4hyf.json.gz
4hyf.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4hyf_validation.pdf.gz
4hyf_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4hyf_full_validation.pdf.gz
4hyf_full_validation.pdf.gz 4hyf_validation.xml.gz
4hyf_validation.xml.gz 4hyf_validation.cif.gz
4hyf_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/hy/4hyf
https://data.pdbj.org/pub/pdb/validation_reports/hy/4hyf ftp://data.pdbj.org/pub/pdb/validation_reports/hy/4hyf
ftp://data.pdbj.org/pub/pdb/validation_reports/hy/4hyf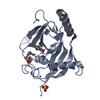
 リンク
リンク 集合体
集合体



 要素
要素 Homo sapiens (ヒト) / 遺伝子: PARP5B, TANK2, TNKL, TNKS2 / プラスミド: PNIC-BSA4 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PARP5B, TANK2, TNKL, TNKS2 / プラスミド: PNIC-BSA4 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 1.28243 Å
/ ビームライン: X06DA / 波長: 1.28243 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj