| 登録情報 | データベース: PDB / ID: 4d6o
|
|---|
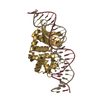
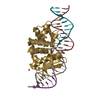
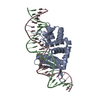
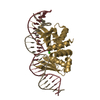
| タイトル | THE CRYSTAL STRUCTURE OF I-DMOI IN COMPLEX WITH ITS TARGET DNA AT 1H INCUBATION IN 5MM MG (STATE 2) |
|---|
 要素 要素 | - (25MER) x 4
- HOMING ENDONUCLEASE I-DMOI
|
|---|
 キーワード キーワード | HYDROLASE/DNA / HYDROLASE-DNA COMPLEX / CATALYSIS / PROTEIN-DNA INTERACTION / X-RAY CRYSTALLOGRAPHY. |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
intron homing / intein-mediated protein splicing / endonuclease activity / 加水分解酵素; エステル加水分解酵素類似検索 - 分子機能 LAGLIDADG-like domain / Intein / Homing endonucleases / Endonuclease I-creI / Intein DOD homing endonuclease / Intein DOD-type homing endonuclease domain profile. / Homing endonuclease, LAGLIDADG / Homing endonuclease / Roll / Alpha Beta類似検索 - ドメイン・相同性 ACETATE ION / DNA / DNA (> 10) / Homing endonuclease I-DmoI類似検索 - 構成要素 |
|---|
| 生物種 |  DESULFUROCOCCUS MOBILIS (古細菌) DESULFUROCOCCUS MOBILIS (古細菌)
SYNTHETIC CONSTRUCT (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å |
|---|
 データ登録者 データ登録者 | Molina, R. / Stella, S. / Redondo, P. / Gomez, H. / Marcaida, M.J. / Orozco, M. / Prieto, J. / Montoya, G. |
|---|
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2015 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2015
タイトル: Visualizing Phosphodiester-Bond Hydrolysis by an Endonuclease.
著者: Molina, R. / Stella, S. / Redondo, P. / Gomez, H. / Marcaida, M.J. / Orozco, M. / Prieto, J. / Montoya, G. |
|---|
| 履歴 | | 登録 | 2014年11月13日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2014年12月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年1月21日 | Group: Database references |
|---|
| 改定 1.2 | 2017年8月23日 | Group: Data collection / カテゴリ: diffrn_detector / Item: _diffrn_detector.type |
|---|
| 改定 1.3 | 2023年12月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 DESULFUROCOCCUS MOBILIS (古細菌)
DESULFUROCOCCUS MOBILIS (古細菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2015
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4d6o.cif.gz
4d6o.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4d6o.ent.gz
pdb4d6o.ent.gz PDB形式
PDB形式 4d6o.json.gz
4d6o.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4d6o_validation.pdf.gz
4d6o_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4d6o_full_validation.pdf.gz
4d6o_full_validation.pdf.gz 4d6o_validation.xml.gz
4d6o_validation.xml.gz 4d6o_validation.cif.gz
4d6o_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/d6/4d6o
https://data.pdbj.org/pub/pdb/validation_reports/d6/4d6o ftp://data.pdbj.org/pub/pdb/validation_reports/d6/4d6o
ftp://data.pdbj.org/pub/pdb/validation_reports/d6/4d6o リンク
リンク 集合体
集合体



 要素
要素 DESULFUROCOCCUS MOBILIS (古細菌) / プラスミド: PET24D / 発現宿主:
DESULFUROCOCCUS MOBILIS (古細菌) / プラスミド: PET24D / 発現宿主: 





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 0.98
/ ビームライン: X06DA / 波長: 0.98  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj









































