+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4ceu | ||||||
|---|---|---|---|---|---|---|---|
| Title | 1.58 A resolution native Sporosarcina pasteurii urease | ||||||
 Components Components | (UREASE SUBUNIT ...) x 3 | ||||||
 Keywords Keywords | HYDROLASE | ||||||
| Function / homology |  Function and homology information Function and homology informationurease complex / urease / urease activity / urea catabolic process / nickel cation binding / cytoplasm Similarity search - Function | ||||||
| Biological species |  SPOROSARCINA PASTEURII (bacteria) SPOROSARCINA PASTEURII (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.58 Å MOLECULAR REPLACEMENT / Resolution: 1.58 Å | ||||||
 Authors Authors | Benini, S. / Cianci, M. / Ciurli, S. | ||||||
 Citation Citation |  Journal: J.Biol.Inorg.Chem. / Year: 2014 Journal: J.Biol.Inorg.Chem. / Year: 2014Title: Fluoride Inhibition of Sporosarcina Pasteurii Urease: Structure and Thermodynamics. Authors: Benini, S. / Cianci, M. / Mazzei, L. / Ciurli, S. #1:  Journal: J.Biol.Inorg.Chem. / Year: 2013 Journal: J.Biol.Inorg.Chem. / Year: 2013Title: The Crystal Structure of Sporosarcina Pasteurii Urease in a Complex with Citrate Provides New Hints for Inhibitor Design. Authors: Benini, S. / Kosikowska, P. / Cianci, M. / Mazzei, L. / Vara, A.G. / Berlicki, L. / Ciurli, S. #2:  Journal: J.Am.Chem.Soc. / Year: 2004 Journal: J.Am.Chem.Soc. / Year: 2004Title: Molecular Details of Urease Inhibition by Boric Acid: Insights Into the Catalytic Mechanism. Authors: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Mangani, S. / Ciurli, S. #3:  Journal: J.Biol.Inorg.Chem. / Year: 2001 Journal: J.Biol.Inorg.Chem. / Year: 2001Title: Structure-Based Rationalization of Urease Inhibition by Phosphate: Novel Insights Into the Enzyme Mechanism. Authors: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Ciurli, S. / Mangani, S. #4:  Journal: J.Biol.Inorg.Chem. / Year: 2000 Journal: J.Biol.Inorg.Chem. / Year: 2000Title: The Complex of Bacillus Pasteurii Urease with Acetohydroxamate Anion from X-Ray Data at 1.55 A Resolution. Authors: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Miletti, S. / Ciurli, S. / Mangani, S. #5:  Journal: Structure / Year: 1999 Journal: Structure / Year: 1999Title: A New Proposal for Urease Mechanism Based on the Crystal Structures of the Native and Inhibited Enzyme from Bacillus Pasteurii: Why Urea Hydrolysis Costs Two Nickels. Authors: Benini, S. / Rypniewski, W.R. / Wilson, K.S. / Miletti, S. / Ciurli, S. / Mangani, S. #6:  Journal: J.Biol.Inorg.Chem. / Year: 1998 Journal: J.Biol.Inorg.Chem. / Year: 1998Title: The Complex of Bacillus Pasteurii Urease with Beta-Mercaptoethanol from X-Ray Data at 1.65 A Resolution Authors: Benini, S. / Ciurli, S. / Rypniewski, W.R. / Wilson, K.S. / Mangani, S. #7: Journal: Acta Crystallogr.,Sect.D / Year: 1998 Title: Crystallization and Preliminary High-Resolution X-Ray Diffraction Analysis of Native and Beta-Mercaptoethanol-Inhibited Urease from Bacillus Pasteurii. Authors: Benini, S. / Ciurli, S. / Rypniewski, W.R. / Wilson, K.S. / Mangani, S. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4ceu.cif.gz 4ceu.cif.gz | 188.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4ceu.ent.gz pdb4ceu.ent.gz | 146.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4ceu.json.gz 4ceu.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ce/4ceu https://data.pdbj.org/pub/pdb/validation_reports/ce/4ceu ftp://data.pdbj.org/pub/pdb/validation_reports/ce/4ceu ftp://data.pdbj.org/pub/pdb/validation_reports/ce/4ceu | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4cexC  4ac7S C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
|
- Components
Components
-UREASE SUBUNIT ... , 3 types, 3 molecules ABC
| #1: Protein | Mass: 11204.988 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Details: GERMAN COLLECTION OF MICROORGANISMS (DSM) / Source: (natural)  SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41022, urease SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41022, urease |
|---|---|
| #2: Protein | Mass: 13975.539 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Details: GERMAN COLLECTION OF MICROORGANISMS (DSM) / Source: (natural)  SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41021, urease SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41021, urease |
| #3: Protein | Mass: 61646.766 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Details: GERMAN COLLECTION OF MICROORGANISMS (DSM) / Source: (natural)  SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41020, urease SPOROSARCINA PASTEURII (bacteria) / Strain: DSM33 / References: UniProt: P41020, urease |
-Non-polymers , 5 types, 879 molecules 


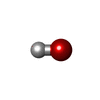





| #4: Chemical | ChemComp-EDO / #5: Chemical | ChemComp-SO4 / #6: Chemical | #7: Chemical | ChemComp-OH / | #8: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.73 Å3/Da / Density % sol: 55.01 % / Description: NONE |
|---|---|
| Crystal grow | pH: 6.5 Details: 1.8 M AMMONIUM SULFATE, 100 MM CITRATE, PH 6.3, 50 MM NA2SO3 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / Beamline: P13 (MX1) / Wavelength: 0.968 / Beamline: P13 (MX1) / Wavelength: 0.968 |
| Detector | Type: MARRESEARCH / Detector: CCD / Date: Jun 18, 2012 |
| Radiation | Monochromator: SI(III) CRYSTAL MONOCHROMATOR / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.968 Å / Relative weight: 1 |
| Reflection | Resolution: 1.58→97.51 Å / Num. obs: 130522 / % possible obs: 100 % / Observed criterion σ(I): 2 / Redundancy: 9.8 % / Rmerge(I) obs: 0.09 / Net I/σ(I): 23 |
| Reflection shell | Resolution: 1.58→1.62 Å / Redundancy: 8.8 % / Rmerge(I) obs: 0.52 / Mean I/σ(I) obs: 4.5 / % possible all: 100 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 4AC7 Resolution: 1.58→97.51 Å / Cor.coef. Fo:Fc: 0.976 / Cor.coef. Fo:Fc free: 0.97 / SU B: 1.035 / SU ML: 0.037 / Cross valid method: THROUGHOUT / ESU R: 0.059 / ESU R Free: 0.059 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 15.176 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.58→97.51 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj








