| 登録情報 | データベース: PDB / ID: 4bdw
|
|---|
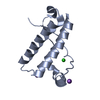
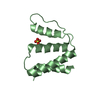
| タイトル | The structure of the FnI-EGF tandem domain of coagulation factor XII in complex with Holmium |
|---|
 要素 要素 | COAGULATION FACTOR XIIA HEAVY CHAIN |
|---|
 キーワード キーワード | HYDROLASE / FNI DOMAIN / EGF DOMAIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
coagulation factor XIIa / plasma kallikrein-kinin cascade / Factor XII activation / Defective SERPING1 causes hereditary angioedema / response to misfolded protein / positive regulation of plasminogen activation / blood coagulation, intrinsic pathway / positive regulation of fibrinolysis / misfolded protein binding / zymogen activation ...coagulation factor XIIa / plasma kallikrein-kinin cascade / Factor XII activation / Defective SERPING1 causes hereditary angioedema / response to misfolded protein / positive regulation of plasminogen activation / blood coagulation, intrinsic pathway / positive regulation of fibrinolysis / misfolded protein binding / zymogen activation / Defective factor XII causes hereditary angioedema / protein autoprocessing / positive regulation of blood coagulation / fibrinolysis / rough endoplasmic reticulum / Intrinsic Pathway of Fibrin Clot Formation / protein processing / blood coagulation / : / serine-type endopeptidase activity / innate immune response / calcium ion binding / extracellular space / extracellular exosome / extracellular region / plasma membrane類似検索 - 分子機能 Coagulation factor XII/hepatocyte growth factor activator / Fibronectin type I domain / Fibronectin, type I / Fibronectin type-I domain signature. / Fibronectin type-I domain profile. / Fibronectin type 1 domain / Fibronectin type II domain / Fibronectin type II domain superfamily / Fibronectin type II domain / Fibronectin type-II collagen-binding domain signature. ...Coagulation factor XII/hepatocyte growth factor activator / Fibronectin type I domain / Fibronectin, type I / Fibronectin type-I domain signature. / Fibronectin type-I domain profile. / Fibronectin type 1 domain / Fibronectin type II domain / Fibronectin type II domain superfamily / Fibronectin type II domain / Fibronectin type-II collagen-binding domain signature. / Fibronectin type-II collagen-binding domain profile. / Fibronectin type 2 domain / Laminin / Laminin / EGF-like domain / Kringle domain / Kringle / EGF-like calcium-binding domain / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle domain / Calcium-binding EGF-like domain / : / Kringle-like fold / Epidermal growth factor-like domain. / EGF-like domain profile. / EGF-like domain signature 1. / EGF-like domain signature 2. / EGF-like domain / Ribbon / Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease / Serine proteases, trypsin domain / Trypsin / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単一同系置換・異常分散 / 解像度: 2.501 Å 単一同系置換・異常分散 / 解像度: 2.501 Å |
|---|
 データ登録者 データ登録者 | Beringer, D.X. / Kroon-Batenburg, L.M.J. |
|---|
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.F / 年: 2013 ジャーナル: Acta Crystallogr.,Sect.F / 年: 2013
タイトル: The Structure of the Fni-Egf-Like Tandem Domain of Coagulation Factor Xii Solved Using Siras.
著者: Beringer, D.X. / Kroon-Batenburg, L.M.J. |
|---|
| 履歴 | | 登録 | 2012年10月8日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2013年2月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年2月20日 | Group: Database references |
|---|
| 改定 1.2 | 2013年4月17日 | Group: Other / Structure summary |
|---|
| 改定 1.3 | 2016年12月28日 | Group: Database references |
|---|
| 改定 1.4 | 2017年7月12日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 2.0 | 2024年11月13日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Database references / Derived calculations / Other / Structure summary
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_entry_details / pdbx_modification_feature / struct_site
Item: _atom_site.occupancy / _database_2.pdbx_DOI ..._atom_site.occupancy / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単一同系置換・異常分散 / 解像度: 2.501 Å
単一同系置換・異常分散 / 解像度: 2.501 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acta Crystallogr.,Sect.F / 年: 2013
ジャーナル: Acta Crystallogr.,Sect.F / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4bdw.cif.gz
4bdw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4bdw.ent.gz
pdb4bdw.ent.gz PDB形式
PDB形式 4bdw.json.gz
4bdw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4bdw_validation.pdf.gz
4bdw_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4bdw_full_validation.pdf.gz
4bdw_full_validation.pdf.gz 4bdw_validation.xml.gz
4bdw_validation.xml.gz 4bdw_validation.cif.gz
4bdw_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bd/4bdw
https://data.pdbj.org/pub/pdb/validation_reports/bd/4bdw ftp://data.pdbj.org/pub/pdb/validation_reports/bd/4bdw
ftp://data.pdbj.org/pub/pdb/validation_reports/bd/4bdw リンク
リンク 集合体
集合体
 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PPICZALPHA / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PPICZALPHA / 発現宿主:  KOMAGATAELLA PASTORIS (菌類) / 株 (発現宿主): X-33 / 参照: UniProt: P00748, coagulation factor XIIa
KOMAGATAELLA PASTORIS (菌類) / 株 (発現宿主): X-33 / 参照: UniProt: P00748, coagulation factor XIIa X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 1.5352
/ ビームライン: X06SA / 波長: 1.5352  解析
解析 単一同系置換・異常分散
単一同系置換・異常分散 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj









