+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3oho | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | catalytic domain of stromelysin-1 in complex with N-Hydroxy-2-(4-methylphenylsulfonamido)acetamide | ||||||
 要素 要素 | Stromelysin-1 | ||||||
 キーワード キーワード | HYDROLASE / Matrixmetalloproteinase | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報stromelysin 1 / cellular response to UV-A / regulation of neuroinflammatory response / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases / response to amyloid-beta / Collagen degradation / collagen catabolic process / negative regulation of reactive oxygen species metabolic process / extracellular matrix disassembly ...stromelysin 1 / cellular response to UV-A / regulation of neuroinflammatory response / Assembly of collagen fibrils and other multimeric structures / Activation of Matrix Metalloproteinases / response to amyloid-beta / Collagen degradation / collagen catabolic process / negative regulation of reactive oxygen species metabolic process / extracellular matrix disassembly / Degradation of the extracellular matrix / extracellular matrix organization / EGFR Transactivation by Gastrin / extracellular matrix / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / cellular response to nitric oxide / regulation of cell migration / cellular response to reactive oxygen species / cellular response to amino acid stimulus / positive regulation of protein-containing complex assembly / protein catabolic process / metalloendopeptidase activity / metallopeptidase activity / peptidase activity / cellular response to lipopolysaccharide / Interleukin-4 and Interleukin-13 signaling / endopeptidase activity / Extra-nuclear estrogen signaling / innate immune response / serine-type endopeptidase activity / mitochondrion / proteolysis / extracellular space / extracellular region / zinc ion binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Kowatz, T. / Naismith, J.H. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: Non-Resonance Raman Difference Spectroscopy as a Tool to Probe Enthalpy-Entropy Compensation and the Interfacial Mobility Model 著者: Kowatz, T. / Naismith, J.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3oho.cif.gz 3oho.cif.gz | 84.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3oho.ent.gz pdb3oho.ent.gz | 63.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3oho.json.gz 3oho.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3oho_validation.pdf.gz 3oho_validation.pdf.gz | 743.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3oho_full_validation.pdf.gz 3oho_full_validation.pdf.gz | 745.4 KB | 表示 | |
| XML形式データ |  3oho_validation.xml.gz 3oho_validation.xml.gz | 8.8 KB | 表示 | |
| CIF形式データ |  3oho_validation.cif.gz 3oho_validation.cif.gz | 10.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/oh/3oho https://data.pdbj.org/pub/pdb/validation_reports/oh/3oho ftp://data.pdbj.org/pub/pdb/validation_reports/oh/3oho ftp://data.pdbj.org/pub/pdb/validation_reports/oh/3oho | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 19002.119 Da / 分子数: 1 / 断片: CATALYTIC DOMAIN (UNP Residues 100-268) / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MMP3, STMY1 / プラスミド: pET28b / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MMP3, STMY1 / プラスミド: pET28b / 発現宿主:  |
|---|
-非ポリマー , 5種, 22分子 

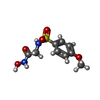






| #2: 化合物 | | #3: 化合物 | #4: 化合物 | ChemComp-Z79 / | #5: 化合物 | ChemComp-SO4 / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.11 Å3/Da / 溶媒含有率: 41.65 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6.5 詳細: 0.1 M (NH4)2SO4, 0.1 M Na-cacodylate, pH 6.5, 26% PEG 8000, VAPOR DIFFUSION, SITTING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.542 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.542 Å |
| 検出器 | タイプ: RIGAKU SATURN 944+ / 検出器: CCD / 日付: 2008年5月2日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.542 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→25.6 Å / Num. obs: 5809 / 冗長度: 3.6 % / Rmerge(I) obs: 0.065 / Net I/σ(I): 26.6 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1b8y 解像度: 2.5→25.6 Å / Cor.coef. Fo:Fc: 0.909 / Cor.coef. Fo:Fc free: 0.885 / SU B: 24.34 / SU ML: 0.266 / 交差検証法: THROUGHOUT / ESU R Free: 0.328 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: BABINET MODEL WITH MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 94.18 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→25.6 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.498→2.563 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj











