| 登録情報 | データベース: PDB / ID: 3o64
|
|---|
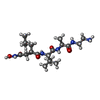
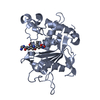
| タイトル | Crystal structure of catalytic domain of TACE with 2-(2-Aminothiazol-4-yl)pyrrolidine-Based Tartrate Diamides |
|---|
 要素 要素 | TACE |
|---|
 キーワード キーワード | hydrolase/hydrolase inhibitor / Hydrolase / ADAM Proteins / Enzyme Inhibitors / hydrolase-hydrolase inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
ADAM 17 endopeptidase / regulation of mast cell apoptotic process / : / signal release / metalloendopeptidase activity involved in amyloid precursor protein catabolic process / cellular response to high density lipoprotein particle stimulus / production of molecular mediator involved in inflammatory response / Constitutive Signaling by NOTCH1 t(7;9)(NOTCH1:M1580_K2555) Translocation Mutant / Notch receptor processing / tumor necrosis factor binding ...ADAM 17 endopeptidase / regulation of mast cell apoptotic process / : / signal release / metalloendopeptidase activity involved in amyloid precursor protein catabolic process / cellular response to high density lipoprotein particle stimulus / production of molecular mediator involved in inflammatory response / Constitutive Signaling by NOTCH1 t(7;9)(NOTCH1:M1580_K2555) Translocation Mutant / Notch receptor processing / tumor necrosis factor binding / interleukin-6 receptor binding / positive regulation of T cell chemotaxis / TNF signaling / cytokine precursor processing / positive regulation of leukocyte chemotaxis / regulation of axon regeneration / Release of Hh-Np from the secreting cell / Regulated proteolysis of p75NTR / metallodipeptidase activity / commissural neuron axon guidance / regulation of neuron migration / positive regulation of tumor necrosis factor-mediated signaling pathway / neutrophil mediated immunity / germinal center formation / Notch binding / wound healing, spreading of epidermal cells / positive regulation of vascular endothelial cell proliferation / negative regulation of cold-induced thermogenesis / CD163 mediating an anti-inflammatory response / positive regulation of epidermal growth factor receptor signaling pathway / cell adhesion mediated by integrin / Signaling by EGFR / amyloid precursor protein catabolic process / cytokine binding / Collagen degradation / membrane protein ectodomain proteolysis / positive regulation of blood vessel endothelial cell migration / positive regulation of G1/S transition of mitotic cell cycle / Growth hormone receptor signaling / Nuclear signaling by ERBB4 / spleen development / positive regulation of chemokine production / Notch signaling pathway / Constitutive Signaling by NOTCH1 HD Domain Mutants / Activated NOTCH1 Transmits Signal to the Nucleus / B cell differentiation / PDZ domain binding / phosphatidylinositol 3-kinase/protein kinase B signal transduction / cell motility / negative regulation of transforming growth factor beta receptor signaling pathway / protein processing / metalloendopeptidase activity / SH3 domain binding / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / integrin binding / epidermal growth factor receptor signaling pathway / metallopeptidase activity / positive regulation of tumor necrosis factor production / T cell differentiation in thymus / peptidase activity / actin cytoskeleton / negative regulation of neuron projection development / positive regulation of cell growth / endopeptidase activity / response to lipopolysaccharide / response to hypoxia / cell adhesion / defense response to Gram-positive bacterium / positive regulation of cell migration / apical plasma membrane / membrane raft / endoplasmic reticulum lumen / response to xenobiotic stimulus / Golgi membrane / positive regulation of cell population proliferation / cell surface / proteolysis / metal ion binding / membrane / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 ADAM17, membrane-proximal domain / Membrane-proximal domain, switch, for ADAM17 / Metallo-peptidase family M12 / ADAM10/ADAM17 catalytic domain / : / Disintegrin / Disintegrin domain profile. / Homologues of snake disintegrins / Disintegrin domain / Disintegrin domain superfamily ...ADAM17, membrane-proximal domain / Membrane-proximal domain, switch, for ADAM17 / Metallo-peptidase family M12 / ADAM10/ADAM17 catalytic domain / : / Disintegrin / Disintegrin domain profile. / Homologues of snake disintegrins / Disintegrin domain / Disintegrin domain superfamily / Peptidase M12B, ADAM/reprolysin / ADAM type metalloprotease domain profile. / Collagenase (Catalytic Domain) / Collagenase (Catalytic Domain) / Metallopeptidase, catalytic domain superfamily / Neutral zinc metallopeptidases, zinc-binding region signature. / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 3,N(D,L-[2-(HYDROXYAMINO-CARBONYL)METHYL]-4-METHYL PENTANOYL)L-3-(TERT-BUTYL)GLYCYL-L-ALANINE / Chem-786 / Chem-INN / ISOPROPYL ALCOHOL / Disintegrin and metalloproteinase domain-containing protein 17類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.88 Å 分子置換 / 解像度: 1.88 Å |
|---|
 データ登録者 データ登録者 | Orth, P. |
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2011 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2011
タイトル: 2-(2-Aminothiazol-4-yl)pyrrolidine-based tartrate diamides as potent, selective and orally bioavailable TACE inhibitors.
著者: Dai, C. / Li, D. / Popovici-Muller, J. / Zhao, L. / Girijavallabhan, V.M. / Rosner, K.E. / Lavey, B.J. / Rizvi, R. / Shankar, B.B. / Wong, M.K. / Guo, Z. / Orth, P. / Strickland, C.O. / Sun, ...著者: Dai, C. / Li, D. / Popovici-Muller, J. / Zhao, L. / Girijavallabhan, V.M. / Rosner, K.E. / Lavey, B.J. / Rizvi, R. / Shankar, B.B. / Wong, M.K. / Guo, Z. / Orth, P. / Strickland, C.O. / Sun, J. / Niu, X. / Chen, S. / Kozlowski, J.A. / Lundell, D.J. / Piwinski, J.J. / Shih, N.Y. / Siddiqui, M.A. |
|---|
| 履歴 | | 登録 | 2010年7月28日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2011年4月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2012年12月12日 | Group: Other |
|---|
| 改定 1.3 | 2023年9月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_alt_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_alt_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_ptnr1_label_alt_id / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年11月6日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.88 Å
分子置換 / 解像度: 1.88 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2011
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3o64.cif.gz
3o64.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3o64.ent.gz
pdb3o64.ent.gz PDB形式
PDB形式 3o64.json.gz
3o64.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3o64_validation.pdf.gz
3o64_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3o64_full_validation.pdf.gz
3o64_full_validation.pdf.gz 3o64_validation.xml.gz
3o64_validation.xml.gz 3o64_validation.cif.gz
3o64_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o6/3o64
https://data.pdbj.org/pub/pdb/validation_reports/o6/3o64 ftp://data.pdbj.org/pub/pdb/validation_reports/o6/3o64
ftp://data.pdbj.org/pub/pdb/validation_reports/o6/3o64
 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: ADAM17, CSVP, TACE / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ADAM17, CSVP, TACE / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P78536, ADAM 17 endopeptidase
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P78536, ADAM 17 endopeptidase









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CHESS
CHESS  / ビームライン: A1 / 波長: 0.9766 Å
/ ビームライン: A1 / 波長: 0.9766 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj
















