+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3nfl | ||||||
|---|---|---|---|---|---|---|---|
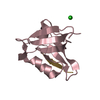
| タイトル | Crystal structure of the PTPN4 PDZ domain complexed with the C-terminus of the GluN2A NMDA receptor subunit | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PROTEIN BINDING / PDZ-PDZ-binding site complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報excitatory chemical synaptic transmission / directional locomotion / Toll Like Receptor 4 (TLR4) Cascade / Synaptic adhesion-like molecules / serotonin metabolic process / protein localization to postsynaptic membrane / sleep / Interleukin-37 signaling / regulation of monoatomic cation transmembrane transport / Assembly and cell surface presentation of NMDA receptors ...excitatory chemical synaptic transmission / directional locomotion / Toll Like Receptor 4 (TLR4) Cascade / Synaptic adhesion-like molecules / serotonin metabolic process / protein localization to postsynaptic membrane / sleep / Interleukin-37 signaling / regulation of monoatomic cation transmembrane transport / Assembly and cell surface presentation of NMDA receptors / NMDA glutamate receptor activity / NMDA selective glutamate receptor complex / Neurexins and neuroligins / glutamate receptor signaling pathway / calcium ion transmembrane import into cytosol / startle response / monoatomic cation transmembrane transport / dopamine metabolic process / Negative regulation of NMDA receptor-mediated neuronal transmission / Unblocking of NMDA receptors, glutamate binding and activation / non-membrane spanning protein tyrosine phosphatase activity / Long-term potentiation / regulation of neuronal synaptic plasticity / positive regulation of excitatory postsynaptic potential / glutamate receptor binding / positive regulation of synaptic transmission, glutamatergic / protein dephosphorylation / MECP2 regulates neuronal receptors and channels / cytoskeletal protein binding / glutamate-gated calcium ion channel activity / neurogenesis / sensory perception of pain / protein-tyrosine-phosphatase / sodium ion transmembrane transport / ionotropic glutamate receptor signaling pathway / cytoplasmic vesicle membrane / protein tyrosine phosphatase activity / synaptic membrane / response to amphetamine / excitatory postsynaptic potential / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / synaptic transmission, glutamatergic / protein catabolic process / postsynaptic density membrane / negative regulation of protein catabolic process / brain development / calcium ion transmembrane transport / visual learning / regulation of synaptic plasticity / response to wounding / memory / cytoplasmic side of plasma membrane / long-term synaptic potentiation / synaptic vesicle / amyloid-beta binding / presynaptic membrane / response to ethanol / chemical synaptic transmission / dendritic spine / postsynaptic membrane / learning or memory / cytoskeleton / postsynaptic density / positive regulation of apoptotic process / response to xenobiotic stimulus / endoplasmic reticulum membrane / glutamatergic synapse / cell surface / zinc ion binding / nucleoplasm / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.91 Å 分子置換 / 解像度: 1.91 Å | ||||||
 データ登録者 データ登録者 | Babault, N. / Cordier, F. / Lafage, M. / Cockburn, J. / Haouz, A. / Rey, F.A. / Delepierre, M. / Buc, H. / Lafon, M. / Wolff, N. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2011 ジャーナル: Structure / 年: 2011タイトル: Peptides Targeting the PDZ Domain of PTPN4 Are Efficient Inducers of Glioblastoma Cell Death. 著者: Babault, N. / Cordier, F. / Lafage, M. / Cockburn, J. / Haouz, A. / Prehaud, C. / Rey, F.A. / Delepierre, M. / Buc, H. / Lafon, M. / Wolff, N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3nfl.cif.gz 3nfl.cif.gz | 166.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3nfl.ent.gz pdb3nfl.ent.gz | 133.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3nfl.json.gz 3nfl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3nfl_validation.pdf.gz 3nfl_validation.pdf.gz | 469.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3nfl_full_validation.pdf.gz 3nfl_full_validation.pdf.gz | 471.5 KB | 表示 | |
| XML形式データ |  3nfl_validation.xml.gz 3nfl_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  3nfl_validation.cif.gz 3nfl_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nf/3nfl https://data.pdbj.org/pub/pdb/validation_reports/nf/3nfl ftp://data.pdbj.org/pub/pdb/validation_reports/nf/3nfl ftp://data.pdbj.org/pub/pdb/validation_reports/nf/3nfl | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11694.363 Da / 分子数: 4 / 断片: PDZ domain (UNP residues 499-604) / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PTPN4 / プラスミド: pDEST15 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PTPN4 / プラスミド: pDEST15 / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 1913.204 Da / 分子数: 4 / 断片: UNP residues 1449-1464 / 由来タイプ: 合成 詳細: NR2A16 peptide has been chemically synthetized. The sequence ocuurs naturally in Homo sapiens (Human) 由来: (合成)  Homo sapiens (ヒト) / 参照: UniProt: Q12879 Homo sapiens (ヒト) / 参照: UniProt: Q12879#3: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.34 Å3/Da / 溶媒含有率: 47.38 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 24% PEG 4000, 0.2M ammonium sulfate, VAPOR DIFFUSION, HANGING DROP, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 0.9795 Å / ビームライン: X06SA / 波長: 0.9795 Å |
| 検出器 | タイプ: PSI PILATUS 6M / 検出器: PIXEL / 日付: 2009年8月8日 |
| 放射 | モノクロメーター: LN2 cooled fixed-exit Si(111) Dynamically bendable mirror プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9795 Å / 相対比: 1 |
| 反射 | 解像度: 1.91→95.45 Å / Num. all: 40560 / Num. obs: 38978 / % possible obs: 96.1 % / 冗長度: 4.2 % / Biso Wilson estimate: 37.4 Å2 / Rmerge(I) obs: 0.043 / Net I/σ(I): 21.1 |
| 反射 シェル | 解像度: 1.91→2.01 Å / 冗長度: 2.1 % / Rmerge(I) obs: 0.309 / Mean I/σ(I) obs: 2 / Num. unique all: 4287 / % possible all: 74.7 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 2VPH 解像度: 1.91→49.88 Å / Cor.coef. Fo:Fc: 0.9235 / Cor.coef. Fo:Fc free: 0.9025 / SU R Cruickshank DPI: 0.144 / 交差検証法: THROUGHOUT / σ(F): 0
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 54.07 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.387 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.91→49.88 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.91→1.96 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj
















