| 登録情報 | データベース: PDB / ID: 3n1r
|
|---|
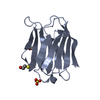
| タイトル | Crystal Structure of ShhN |
|---|
 要素 要素 | Sonic hedgehog protein |
|---|
 キーワード キーワード | PROTEIN BINDING / Binding Sites / Calcium / Cell Adhesion Molecules / Cell Cycle Proteins / Cell Line / Conserved Sequence / Fibronectins / Hedgehog Proteins / Immunoglobulin G / Membrane Glycoproteins / Membrane Proteins / Tertiary / Receptors / Cell Surface / Sequence Homology / Signal Transduction / Tumor Suppressor Proteins |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
forebrain regionalization / cell proliferation in external granule layer / zona limitans intrathalamica formation / positive regulation of neurotrophin production / positive regulation of photoreceptor cell differentiation / fungiform papilla development / Release of Hh-Np from the secreting cell / digestive tract mesoderm development / epithelial-mesenchymal signaling involved in prostate gland development / fungiform papilla morphogenesis ...forebrain regionalization / cell proliferation in external granule layer / zona limitans intrathalamica formation / positive regulation of neurotrophin production / positive regulation of photoreceptor cell differentiation / fungiform papilla development / Release of Hh-Np from the secreting cell / digestive tract mesoderm development / epithelial-mesenchymal signaling involved in prostate gland development / fungiform papilla morphogenesis / ventral spinal cord interneuron specification / tongue morphogenesis / respiratory tube development / Ligand-receptor interactions / Activation of SMO / mesenchymal-epithelial cell signaling involved in prostate gland development / positive regulation of oligodendrocyte progenitor proliferation / trachea development / positive regulation of skeletal muscle cell proliferation / anatomical structure formation involved in morphogenesis / right lung development / left lung development / primary prostatic bud elongation / regulation of mesenchymal cell proliferation involved in prostate gland development / mesenchymal smoothened signaling pathway involved in prostate gland development / positive regulation of sclerotome development / tracheoesophageal septum formation / negative regulation of ureter smooth muscle cell differentiation / positive regulation of ureter smooth muscle cell differentiation / negative regulation of kidney smooth muscle cell differentiation / positive regulation of kidney smooth muscle cell differentiation / regulation of odontogenesis / positive regulation of mesenchymal cell proliferation involved in ureter development / polarity specification of anterior/posterior axis / trunk neural crest cell migration / hindgut morphogenesis / striated muscle tissue development / negative regulation of alpha-beta T cell differentiation / regulation of glial cell proliferation / regulation of prostatic bud formation / metanephric mesenchymal cell proliferation involved in metanephros development / positive regulation of penile erection / formation of anatomical boundary / lung epithelium development / positive regulation of striated muscle cell differentiation / neural tube formation / trachea morphogenesis / myotube differentiation / regulation of epithelial cell proliferation involved in prostate gland development / cholesterol-protein transferase activity / bud outgrowth involved in lung branching / telencephalon regionalization / epithelial-mesenchymal cell signaling / laminin-1 binding / vasculogenesis involved in coronary vascular morphogenesis / Hedgehog ligand biogenesis / salivary gland cavitation / negative regulation of cholesterol efflux / determination of left/right asymmetry in lateral mesoderm / spinal cord dorsal/ventral patterning / negative regulation of mesenchymal cell apoptotic process / cell development / positive regulation of cerebellar granule cell precursor proliferation / negative regulation of T cell differentiation in thymus / spinal cord motor neuron differentiation / positive regulation of T cell differentiation in thymus / intermediate filament organization / cerebellar granule cell precursor proliferation / mesenchymal cell apoptotic process / embryonic skeletal system development / male genitalia morphogenesis / limb bud formation / lung lobe morphogenesis / prostate gland development / skeletal muscle fiber differentiation / establishment of epithelial cell polarity / fungiform papilla formation / thalamus development / embryonic digestive tract morphogenesis / embryonic foregut morphogenesis / somite development / patched binding / epithelial cell proliferation involved in salivary gland morphogenesis / hindbrain development / positive regulation of skeletal muscle tissue development / animal organ formation / ectoderm development / neuron fate commitment / branching involved in prostate gland morphogenesis / stem cell development / dorsal/ventral neural tube patterning / negative regulation of dopaminergic neuron differentiation / mesenchymal cell proliferation involved in lung development / negative thymic T cell selection / skeletal muscle cell proliferation / lymphoid progenitor cell differentiation / mesenchymal cell proliferation / positive regulation of immature T cell proliferation in thymus / CD4-positive or CD8-positive, alpha-beta T cell lineage commitment / embryonic morphogenesis類似検索 - 分子機能 Hedgehog, N-terminal signalling domain / Hedgehog protein / Hedgehog protein, Hint domain / Hint module / Hedgehog amino-terminal signalling domain / Muramoyl-pentapeptide Carboxypeptidase; domain 2 - #10 / Muramoyl-pentapeptide Carboxypeptidase; domain 2 / Hedgehog signalling/DD-peptidase zinc-binding domain superfamily / Hint domain C-terminal / Hint (Hedgehog/Intein) domain C-terminal region ...Hedgehog, N-terminal signalling domain / Hedgehog protein / Hedgehog protein, Hint domain / Hint module / Hedgehog amino-terminal signalling domain / Muramoyl-pentapeptide Carboxypeptidase; domain 2 - #10 / Muramoyl-pentapeptide Carboxypeptidase; domain 2 / Hedgehog signalling/DD-peptidase zinc-binding domain superfamily / Hint domain C-terminal / Hint (Hedgehog/Intein) domain C-terminal region / Intein N-terminal splicing region / Intein N-terminal splicing motif profile. / Hint domain N-terminal / Hint (Hedgehog/Intein) domain N-terminal region / Hint domain superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.13 Å 分子置換 / 解像度: 2.13 Å |
|---|
 データ登録者 データ登録者 | Kavran, J.M. / Leahy, D.J. |
|---|
 引用 引用 |  ジャーナル: TO BE PUBLISHED ジャーナル: TO BE PUBLISHED
タイトル: Crystal Structure of ShhN
著者: Kavran, J.M. / Ward, M.D. / Oladosu, O.O. / Mulepati, S. / Leahy, D.J. |
|---|
| 履歴 | | 登録 | 2010年5月16日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2010年6月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2024年2月21日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  分子置換 / 解像度: 2.13 Å
分子置換 / 解像度: 2.13 Å  データ登録者
データ登録者 引用
引用 ジャーナル: TO BE PUBLISHED
ジャーナル: TO BE PUBLISHED 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3n1r.cif.gz
3n1r.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3n1r.ent.gz
pdb3n1r.ent.gz PDB形式
PDB形式 3n1r.json.gz
3n1r.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3n1r_validation.pdf.gz
3n1r_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3n1r_full_validation.pdf.gz
3n1r_full_validation.pdf.gz 3n1r_validation.xml.gz
3n1r_validation.xml.gz 3n1r_validation.cif.gz
3n1r_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n1/3n1r
https://data.pdbj.org/pub/pdb/validation_reports/n1/3n1r ftp://data.pdbj.org/pub/pdb/validation_reports/n1/3n1r
ftp://data.pdbj.org/pub/pdb/validation_reports/n1/3n1r リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT
回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT 解析
解析 分子置換 / 解像度: 2.13→27.282 Å / SU ML: 0.34 / σ(F): 1.34 / 立体化学のターゲット値: ML
分子置換 / 解像度: 2.13→27.282 Å / SU ML: 0.34 / σ(F): 1.34 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj









