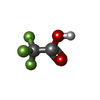
| 登録情報 | データベース: PDB / ID: 3kw9
|
|---|
| タイトル | X-ray structure of Cathepsin K covalently bound to a triazine ligand |
|---|
 要素 要素 | Cathepsin K |
|---|
 キーワード キーワード | HYDROLASE / cysteine / thioimidate / Disulfide bond / cys protease / inhibitor / non-peptide / Protease / Thiol protease / Zymogen |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cathepsin K / negative regulation of cartilage development / RUNX1 regulates transcription of genes involved in differentiation of keratinocytes / endolysosome lumen / thyroid hormone generation / Trafficking and processing of endosomal TLR / proteoglycan binding / Activation of Matrix Metalloproteinases / Collagen degradation / collagen catabolic process ...cathepsin K / negative regulation of cartilage development / RUNX1 regulates transcription of genes involved in differentiation of keratinocytes / endolysosome lumen / thyroid hormone generation / Trafficking and processing of endosomal TLR / proteoglycan binding / Activation of Matrix Metalloproteinases / Collagen degradation / collagen catabolic process / fibronectin binding / extracellular matrix disassembly / bone resorption / mitophagy / collagen binding / Degradation of the extracellular matrix / cysteine-type peptidase activity / MHC class II antigen presentation / lysosomal lumen / proteolysis involved in protein catabolic process / lysosome / apical plasma membrane / serine-type endopeptidase activity / external side of plasma membrane / cysteine-type endopeptidase activity / intracellular membrane-bounded organelle / proteolysis / extracellular space / extracellular region / nucleoplasm類似検索 - 分子機能 Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Papain-like cysteine endopeptidase / Cysteine peptidase, asparagine active site / Eukaryotic thiol (cysteine) proteases asparagine active site. / Cysteine peptidase, histidine active site / Eukaryotic thiol (cysteine) proteases histidine active site. / : / Peptidase C1A, papain C-terminal ...Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Cathepsin propeptide inhibitor domain (I29) / Papain-like cysteine endopeptidase / Cysteine peptidase, asparagine active site / Eukaryotic thiol (cysteine) proteases asparagine active site. / Cysteine peptidase, histidine active site / Eukaryotic thiol (cysteine) proteases histidine active site. / : / Peptidase C1A, papain C-terminal / Papain family cysteine protease / Papain family cysteine protease / Cysteine proteinases / Cysteine peptidase, cysteine active site / Eukaryotic thiol (cysteine) proteases cysteine active site. / Cathepsin B; Chain A / Papain-like cysteine peptidase superfamily / Alpha-Beta Complex / Alpha Beta類似検索 - ドメイン・相同性 Chem-ORG / trifluoroacetic acid / Cathepsin K類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å |
|---|
 データ登録者 データ登録者 | Uitdehaag, J.C.M. / van Zeeland, M. |
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010
タイトル: Design and optimization of a series of novel 2-cyano-pyrimidines as cathepsin K inhibitors.
著者: Rankovic, Z. / Cai, J. / Kerr, J. / Fradera, X. / Robinson, J. / Mistry, A. / Hamilton, E. / McGarry, G. / Andrews, F. / Caulfield, W. / Cumming, I. / Dempster, M. / Waller, J. / Scullion, P. ...著者: Rankovic, Z. / Cai, J. / Kerr, J. / Fradera, X. / Robinson, J. / Mistry, A. / Hamilton, E. / McGarry, G. / Andrews, F. / Caulfield, W. / Cumming, I. / Dempster, M. / Waller, J. / Scullion, P. / Martin, I. / Mitchell, A. / Long, C. / Baugh, M. / Westwood, P. / Kinghorn, E. / Bruin, J. / Hamilton, W. / Uitdehaag, J. / van Zeeland, M. / Potin, D. / Saniere, L. / Fouquet, A. / Chevallier, F. / Deronzier, H. / Dorleans, C. / Nicolai, E. |
|---|
| 履歴 | | 登録 | 2009年12月1日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2010年3月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2013年6月19日 | Group: Database references |
|---|
| 改定 1.3 | 2017年11月1日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.4 | 2024年11月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.8 Å
分子置換 / 解像度: 1.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3kw9.cif.gz
3kw9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3kw9.ent.gz
pdb3kw9.ent.gz PDB形式
PDB形式 3kw9.json.gz
3kw9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3kw9_validation.pdf.gz
3kw9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3kw9_full_validation.pdf.gz
3kw9_full_validation.pdf.gz 3kw9_validation.xml.gz
3kw9_validation.xml.gz 3kw9_validation.cif.gz
3kw9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/kw/3kw9
https://data.pdbj.org/pub/pdb/validation_reports/kw/3kw9 ftp://data.pdbj.org/pub/pdb/validation_reports/kw/3kw9
ftp://data.pdbj.org/pub/pdb/validation_reports/kw/3kw9 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CTSK, CTSO, CTSO2 / 器官 (発現宿主): ovary
Homo sapiens (ヒト) / 遺伝子: CTSK, CTSO, CTSO2 / 器官 (発現宿主): ovary
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-4 / 波長: 0.931 Å
/ ビームライン: ID14-4 / 波長: 0.931 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 1.8→50 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.816 / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber
分子置換 / 解像度: 1.8→50 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.816 / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj