+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3iyp | ||||||
|---|---|---|---|---|---|---|---|
| Title | The Interaction of Decay-accelerating Factor with Echovirus 7 | ||||||
 Components Components |
| ||||||
 Keywords Keywords | VIRUS / RECEPTOR / COMPLEX / ECHOVIRUS / DAF / icosahedral virus | ||||||
| Function / homology |  Function and homology information Function and homology informationregulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / ficolin-1-rich granule membrane ...regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / ficolin-1-rich granule membrane / complement activation, classical pathway / transport vesicle / side of membrane / COPI-mediated anterograde transport / endoplasmic reticulum-Golgi intermediate compartment membrane / secretory granule membrane / Regulation of Complement cascade / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / ribonucleoside triphosphate phosphatase activity / positive regulation of T cell cytokine production / host cell cytoplasmic vesicle membrane / virion component / viral capsid / nucleoside-triphosphate phosphatase / host cell / channel activity / positive regulation of cytosolic calcium ion concentration / virus receptor activity / monoatomic ion transmembrane transport / DNA replication / RNA helicase activity / membrane raft / endocytosis involved in viral entry into host cell / symbiont-mediated suppression of host gene expression / Golgi membrane / innate immune response / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / Neutrophil degranulation / symbiont entry into host cell / lipid binding / virion attachment to host cell / host cell nucleus / structural molecule activity / cell surface / proteolysis / RNA binding / extracellular exosome / extracellular region / zinc ion binding / ATP binding / membrane / plasma membrane Similarity search - Function | ||||||
| Biological species |  Human echovirus 7 Human echovirus 7 Homo sapiens (human) Homo sapiens (human) | ||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 7.2 Å | ||||||
 Authors Authors | Plevka, P. / Hafenstein, S. / Zhang, Y. / Harris, K.G. / Cifuente, J.O. / Bowman, V.D. / Chipman, P.R. / Lin, F. / Medof, D.E. / Bator, C.M. / Rossmann, M.G. | ||||||
 Citation Citation |  Journal: J Virol / Year: 2010 Journal: J Virol / Year: 2010Title: Interaction of decay-accelerating factor with echovirus 7. Authors: Pavel Plevka / Susan Hafenstein / Katherine G Harris / Javier O Cifuente / Ying Zhang / Valorie D Bowman / Paul R Chipman / Carol M Bator / Feng Lin / M Edward Medof / Michael G Rossmann /  Abstract: Echovirus 7 (EV7) belongs to the Enterovirus genus within the family Picornaviridae. Many picornaviruses use IgG-like receptors that bind in the viral canyon and are required to initiate viral ...Echovirus 7 (EV7) belongs to the Enterovirus genus within the family Picornaviridae. Many picornaviruses use IgG-like receptors that bind in the viral canyon and are required to initiate viral uncoating during infection. However, in addition, some of the enteroviruses use an alternative or additional receptor that binds outside the canyon. Decay-accelerating factor (DAF) has been identified as a cellular receptor for EV7. The crystal structure of EV7 has been determined to 3.1-Å resolution and used to interpret the 7.2-Å-resolution cryo-electron microscopy reconstruction of EV7 complexed with DAF. Each DAF binding site on EV7 is near a 2-fold icosahedral symmetry axis, which differs from the binding site of DAF on the surface of coxsackievirus B3, indicating that there are independent evolutionary processes by which DAF was selected as a picornavirus accessory receptor. This suggests that there is an advantage for these viruses to recognize DAF during the initial process of infection. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3iyp.cif.gz 3iyp.cif.gz | 226.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3iyp.ent.gz pdb3iyp.ent.gz | 176.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3iyp.json.gz 3iyp.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  3iyp_validation.pdf.gz 3iyp_validation.pdf.gz | 744.2 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  3iyp_full_validation.pdf.gz 3iyp_full_validation.pdf.gz | 787.8 KB | Display | |
| Data in XML |  3iyp_validation.xml.gz 3iyp_validation.xml.gz | 38.5 KB | Display | |
| Data in CIF |  3iyp_validation.cif.gz 3iyp_validation.cif.gz | 57.3 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/iy/3iyp https://data.pdbj.org/pub/pdb/validation_reports/iy/3iyp ftp://data.pdbj.org/pub/pdb/validation_reports/iy/3iyp ftp://data.pdbj.org/pub/pdb/validation_reports/iy/3iyp | HTTPS FTP |
-Related structure data
| Related structure data |  5179MC  2x5iC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 | x 60
|
| 2 |
|
| 3 | x 5
|
| 4 | x 6
|
| 5 | 
|
| Symmetry | Point symmetry: (Schoenflies symbol: I (icosahedral)) |
- Components
Components
-Protein , 5 types, 5 molecules ABCDF
| #1: Protein | Mass: 33313.203 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Human echovirus 7 / References: UniProt: Q9QP24 Human echovirus 7 / References: UniProt: Q9QP24 |
|---|---|
| #2: Protein | Mass: 26226.959 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Human echovirus 7 / References: UniProt: Q6W9E5 Human echovirus 7 / References: UniProt: Q6W9E5 |
| #3: Protein | Mass: 29072.789 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Human echovirus 7 / References: UniProt: Q6W9E5 Human echovirus 7 / References: UniProt: Q6W9E5 |
| #4: Protein | Mass: 7599.377 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Human echovirus 7 / References: UniProt: Q91QV1 Human echovirus 7 / References: UniProt: Q91QV1 |
| #5: Protein | Mass: 41511.031 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Homo sapiens (human) / References: UniProt: P08174 Homo sapiens (human) / References: UniProt: P08174 |
-Non-polymers , 1 types, 1 molecules 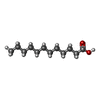
| #6: Chemical | ChemComp-DAO / |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Molecular weight | Value: 6.8 MDa / Experimental value: NO | |||||||||||||||
| Details of virus | Empty: NO / Enveloped: NO / Host category: VERTEBRATES / Isolate: STRAIN / Type: VIRION | |||||||||||||||
| Natural host | Organism: Homo sapiens | |||||||||||||||
| Buffer solution | Name: 0.1 M NaCl, 20mM Tris / pH: 7.2 / Details: 0.1 M NaCl, 20mM Tris | |||||||||||||||
| Specimen | Conc.: 2 mg/ml / Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES / Details: 0.1 M NaCl, 20mM Tris | |||||||||||||||
| Vitrification | Cryogen name: ETHANE / Temp: 113 K / Method: Blot for 2 seconds before plunging Time resolved state: Vitrified 1 hour after mixing DAF with EV7 |
- Electron microscopy imaging
Electron microscopy imaging
| Microscopy | Model: FEI/PHILIPS CM300FEG/T / Date: Jan 1, 2000 Details: Micrographs were digitized with a Zeiss PHODIS microdensitometer at 7-micron intervals. |
|---|---|
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 3670 nm / Nominal defocus min: 1120 nm / Camera length: 0 mm |
| Specimen holder | Specimen holder model: OTHER Specimen holder type: Side entry liquid nitrogen-cooled cryo specimen holder. Temperature: 100 K / Tilt angle max: 0 ° / Tilt angle min: 0 ° |
| Image recording | Electron dose: 20 e/Å2 / Film or detector model: KODAK SO-163 FILM |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Relative weight: 1 |
- Processing
Processing
| EM software |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF correction | Details: Each particle | ||||||||||||||||||
| Symmetry | Point symmetry: I (icosahedral) | ||||||||||||||||||
| 3D reconstruction | Method: common lines fourier method / Resolution: 7.2 Å / Resolution method: FSC 0.5 CUT-OFF / Num. of particles: 11430 / Symmetry type: POINT | ||||||||||||||||||
| Atomic model building |
| ||||||||||||||||||
| Atomic model building | Source name: PDB / Type: experimental model
| ||||||||||||||||||
| Refinement step | Cycle: LAST
|
 Movie
Movie Controller
Controller













 PDBj
PDBj









