+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3hpm | ||||||
|---|---|---|---|---|---|---|---|
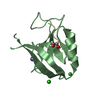
| タイトル | Oxidized dimeric PICK1 PDZ C46G mutant in complex with the carboxyl tail peptide of GluR2 | ||||||
 要素 要素 | PRKCA-binding protein,9-mer peptide of THE GLUR2 SUBUNIT | ||||||
 キーワード キーワード | PROTEIN BINDING / oxidized / PDZ domain | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報G protein-coupled glutamate receptor binding / membrane curvature sensor activity / glial cell development / cellular response to decreased oxygen levels / Arp2/3 complex binding / postsynaptic specialization / regulation of Arp2/3 complex-mediated actin nucleation / postsynaptic endocytic zone / negative regulation of Arp2/3 complex-mediated actin nucleation / dopamine transport ...G protein-coupled glutamate receptor binding / membrane curvature sensor activity / glial cell development / cellular response to decreased oxygen levels / Arp2/3 complex binding / postsynaptic specialization / regulation of Arp2/3 complex-mediated actin nucleation / postsynaptic endocytic zone / negative regulation of Arp2/3 complex-mediated actin nucleation / dopamine transport / SNAP receptor activity / monoamine transport / dendritic spine organization / long-term synaptic depression / dendritic spine maintenance / regulation of postsynaptic neurotransmitter receptor internalization / receptor clustering / Trafficking of GluR2-containing AMPA receptors / positive regulation of receptor internalization / protein targeting / cellular response to glucose starvation / cytoskeletal protein binding / ephrin receptor binding / ionotropic glutamate receptor binding / regulation of insulin secretion / protein kinase C binding / trans-Golgi network membrane / intracellular protein transport / G protein-coupled receptor binding / receptor tyrosine kinase binding / phospholipid binding / actin filament binding / synaptic vesicle / actin binding / presynaptic membrane / GTPase binding / dendritic spine / cytoskeleton / protein phosphorylation / neuron projection / postsynaptic density / signaling receptor binding / protein domain specific binding / negative regulation of gene expression / intracellular membrane-bounded organelle / dendrite / synapse / perinuclear region of cytoplasm / glutamatergic synapse / Golgi apparatus / protein-containing complex / mitochondrion / metal ion binding / identical protein binding / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Yu, J. / Shi, Y. / Zhang, M. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2010 ジャーナル: Biochemistry / 年: 2010タイトル: Redox-Regulated Lipid Membrane Binding of the PICK1 PDZ Domain. 著者: Shi, Y. / Yu, J. / Jia, Y. / Pan, L. / Shen, C. / Xia, J. / Zhang, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3hpm.cif.gz 3hpm.cif.gz | 92.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3hpm.ent.gz pdb3hpm.ent.gz | 70.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3hpm.json.gz 3hpm.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3hpm_validation.pdf.gz 3hpm_validation.pdf.gz | 432.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3hpm_full_validation.pdf.gz 3hpm_full_validation.pdf.gz | 436.9 KB | 表示 | |
| XML形式データ |  3hpm_validation.xml.gz 3hpm_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  3hpm_validation.cif.gz 3hpm_validation.cif.gz | 12.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hp/3hpm https://data.pdbj.org/pub/pdb/validation_reports/hp/3hpm ftp://data.pdbj.org/pub/pdb/validation_reports/hp/3hpm ftp://data.pdbj.org/pub/pdb/validation_reports/hp/3hpm | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Refine code: 2
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 11533.163 Da / 分子数: 2 / 断片: PICK1 PDZ domain / 変異: C46G / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: Pick1, rCG_60080 / プラスミド: pET32a / 発現宿主:  #2: 水 | ChemComp-HOH / | Has protein modification | Y | 配列の詳細 | THIS SEQUENCE IS FUSED WITH PROTEASE 3C CLEAVAGE SITE AND THE C-TERMINAL TAIL 9 AMINO ACID RESIDUES ...THIS SEQUENCE IS FUSED WITH PROTEASE 3C CLEAVAGE SITE AND THE C-TERMINAL TAIL 9 AMINO ACID RESIDUES OF THE GLUR2 SUBUNIT. | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.02 Å3/Da / 溶媒含有率: 39.25 % |
|---|---|
| 結晶化 | 温度: 289 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 20% (w/v) PEG4000, 0.1M HEPES, pH 7.5, VAPOR DIFFUSION, HANGING DROP, temperature 289K |
-データ収集
| 回折 | 平均測定温度: 110 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE |
| 放射 | モノクロメーター: confocal optics / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→46.62 Å / Num. all: 4797 / Num. obs: 4797 / % possible obs: 96.8 % / 冗長度: 4.4 % / Biso Wilson estimate: 77.041 Å2 / Rmerge(I) obs: 0.064 / Net I/σ(I): 7.6 |
| 反射 シェル | 解像度: 2.8→2.95 Å / 冗長度: 4.4 % / Rmerge(I) obs: 0.377 / Mean I/σ(I) obs: 2 / Num. unique all: 683 / % possible all: 96.8 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 2PKU 解像度: 2.8→46.62 Å / Cor.coef. Fo:Fc: 0.919 / Cor.coef. Fo:Fc free: 0.891 / SU B: 46.451 / SU ML: 0.417 / 交差検証法: THROUGHOUT / ESU R Free: 0.519 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 53.494 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→46.62 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Dom-ID: 1 / Auth asym-ID: A / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.872 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj



