+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3g4k | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of human phosphodiesterase 4d with rolipram | ||||||
 要素 要素 | cAMP-specific 3',5'-cyclic phosphodiesterase 4D | ||||||
 キーワード キーワード | HYDROLASE / phosphodiesterase / PDE4D / Alternative splicing / cAMP / Cytoplasm / Cytoskeleton / Membrane / Metal-binding / Phosphoprotein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報signaling receptor regulator activity / negative regulation of relaxation of cardiac muscle / negative regulation of heart contraction / 3',5'-cyclic-AMP phosphodiesterase / positive regulation of interleukin-5 production / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / establishment of endothelial barrier / regulation of cardiac muscle cell contraction / negative regulation of cAMP-mediated signaling / regulation of calcium ion transmembrane transport via high voltage-gated calcium channel ...signaling receptor regulator activity / negative regulation of relaxation of cardiac muscle / negative regulation of heart contraction / 3',5'-cyclic-AMP phosphodiesterase / positive regulation of interleukin-5 production / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / establishment of endothelial barrier / regulation of cardiac muscle cell contraction / negative regulation of cAMP-mediated signaling / regulation of calcium ion transmembrane transport via high voltage-gated calcium channel / voltage-gated calcium channel complex / positive regulation of heart rate / heterocyclic compound binding / adrenergic receptor signaling pathway / regulation of cell communication by electrical coupling involved in cardiac conduction / negative regulation of peptidyl-serine phosphorylation / cAMP catabolic process / 3',5'-cyclic-nucleotide phosphodiesterase activity / DARPP-32 events / calcium channel regulator activity / 3',5'-cyclic-GMP phosphodiesterase activity / 3',5'-cyclic-AMP phosphodiesterase activity / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / beta-2 adrenergic receptor binding / cAMP binding / cellular response to cAMP / calcium channel complex / cellular response to epinephrine stimulus / positive regulation of interleukin-2 production / regulation of heart rate / cAMP-mediated signaling / positive regulation of type II interferon production / T cell receptor signaling pathway / ATPase binding / scaffold protein binding / G alpha (s) signalling events / transmembrane transporter binding / apical plasma membrane / centrosome / perinuclear region of cytoplasm / enzyme binding / membrane / nucleus / metal ion binding / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.95 Å 分子置換 / 解像度: 1.95 Å | ||||||
 データ登録者 データ登録者 | Staker, B.L. | ||||||
 引用 引用 |  ジャーナル: Nat.Biotechnol. / 年: 2010 ジャーナル: Nat.Biotechnol. / 年: 2010タイトル: Design of phosphodiesterase 4D (PDE4D) allosteric modulators for enhancing cognition with improved safety. 著者: Burgin, A.B. / Magnusson, O.T. / Singh, J. / Witte, P. / Staker, B.L. / Bjornsson, J.M. / Thorsteinsdottir, M. / Hrafnsdottir, S. / Hagen, T. / Kiselyov, A.S. / Stewart, L.J. / Gurney, M.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3g4k.cif.gz 3g4k.cif.gz | 303.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3g4k.ent.gz pdb3g4k.ent.gz | 241.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3g4k.json.gz 3g4k.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3g4k_validation.pdf.gz 3g4k_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3g4k_full_validation.pdf.gz 3g4k_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  3g4k_validation.xml.gz 3g4k_validation.xml.gz | 59.4 KB | 表示 | |
| CIF形式データ |  3g4k_validation.cif.gz 3g4k_validation.cif.gz | 87 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/g4/3g4k https://data.pdbj.org/pub/pdb/validation_reports/g4/3g4k ftp://data.pdbj.org/pub/pdb/validation_reports/g4/3g4k ftp://data.pdbj.org/pub/pdb/validation_reports/g4/3g4k | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
| |||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | UNKNOWN |
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 43790.168 Da / 分子数: 4 / 断片: residues 380-753 / 変異: S715A, S717A / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PDE4D, DPDE3 / プラスミド: PBACGUS Homo sapiens (ヒト) / 遺伝子: PDE4D, DPDE3 / プラスミド: PBACGUS発現宿主:  参照: UniProt: Q08499, 3',5'-cyclic-nucleotide phosphodiesterase |
|---|
-非ポリマー , 6種, 1063分子 


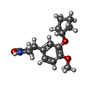







| #2: 化合物 | ChemComp-ZN / #3: 化合物 | ChemComp-MG / #4: 化合物 | #5: 化合物 | ChemComp-ROL / #6: 化合物 | ChemComp-EDO / #7: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.54 Å3/Da / 溶媒含有率: 51.57 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 100MM HEPES PH 7.5, 35% ETHYLENE GLYCOL, 5% GLYCEROL, 22% PEG 3350, VAPOR DIFFUSION, SITTING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 5.0.1 / 波長: 0.97 Å / ビームライン: 5.0.1 / 波長: 0.97 Å |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97 Å / 相対比: 1 |
| 反射 | 解像度: 1.95→50 Å / Num. obs: 131239 / % possible obs: 99.6 % / Rmerge(I) obs: 0.066 / Net I/σ(I): 11.5 |
| 反射 シェル | 解像度: 1.95→1.98 Å / Rmerge(I) obs: 0.699 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.95→50 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.943 / SU B: 3.053 / SU ML: 0.089 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.144 / ESU R Free: 0.133 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS 分子置換 / 解像度: 1.95→50 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.943 / SU B: 3.053 / SU ML: 0.089 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.144 / ESU R Free: 0.133 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.61 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.95→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Ens-ID: 1 / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.95→2 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj








