+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3fl6 | ||||||
|---|---|---|---|---|---|---|---|
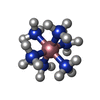
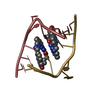
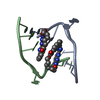
| タイトル | Influence of the incorporation of a cyclohexenyl nucleic acid (CeNA) residue onto the sequence d(GCGTGCG)/d(CGCACGC) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA / double helix / CeNA / sugar modification / right-handed | ||||||
| 機能・相同性 | COBALT HEXAMMINE(III) / DNA 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.17 Å 単波長異常分散 / 解像度: 1.17 Å | ||||||
 データ登録者 データ登録者 | Robeyns, K. / Herdewijn, P. / Van Meervelt, L. | ||||||
 引用 引用 |  ジャーナル: Artif DNA PNA XNA / 年: 2010 ジャーナル: Artif DNA PNA XNA / 年: 2010タイトル: Direct observation of two cyclohexenyl (CeNA) ring conformations in duplex DNA. 著者: Robeyns, K. / Herdewijn, P. / Van Meervelt, L. #1:  ジャーナル: Nucleic Acids Res. / 年: 2008 ジャーナル: Nucleic Acids Res. / 年: 2008タイトル: Influence of the incorporation of a cyclohexenyl nucleic acid (CeNA) residue onto the sequence d(CGCGAATTCGCG). 著者: Robeyns, K. / Herdewijn, P. / Van Meervelt, L. #2:  ジャーナル: J.Am.Chem.Soc. / 年: 2008 ジャーナル: J.Am.Chem.Soc. / 年: 2008タイトル: Structure of the fully modified left-handed cyclohexene nucleic acid sequence GTGTACAC. 著者: Robeyns, K. / Herdewijn, P. / Van Meervelt, L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3fl6.cif.gz 3fl6.cif.gz | 47.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3fl6.ent.gz pdb3fl6.ent.gz | 33.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3fl6.json.gz 3fl6.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3fl6_validation.pdf.gz 3fl6_validation.pdf.gz | 406.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3fl6_full_validation.pdf.gz 3fl6_full_validation.pdf.gz | 411.2 KB | 表示 | |
| XML形式データ |  3fl6_validation.xml.gz 3fl6_validation.xml.gz | 6.1 KB | 表示 | |
| CIF形式データ |  3fl6_validation.cif.gz 3fl6_validation.cif.gz | 7.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fl/3fl6 https://data.pdbj.org/pub/pdb/validation_reports/fl/3fl6 ftp://data.pdbj.org/pub/pdb/validation_reports/fl/3fl6 ftp://data.pdbj.org/pub/pdb/validation_reports/fl/3fl6 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2164.460 Da / 分子数: 2 / 由来タイプ: 合成 #2: DNA鎖 | 分子量: 2083.388 Da / 分子数: 2 / 由来タイプ: 合成 #3: 化合物 | ChemComp-NCO / #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.06 Å3/Da / 溶媒含有率: 40.38 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 289 K / pH: 5.5 詳細: 10%(v/v) 2-methyl-2,4-pentanediol (MPD), 20mM cobalt hexamine, 40mM potassium cacodylate buffered at pH=5.5, and 80/12mM KCl/NaCl, vapor diffusion, hanging drop, temperature 289K | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 0.7749 / ビームライン: X06SA / 波長: 0.7749 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: PSI PILATUS 6M / 検出器: PIXEL / 日付: 2008年4月25日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: UNDULATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 0.7749 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | 冗長度: 8.8 % / Av σ(I) over netI: 2.1 / 数: 71625 / Rmerge(I) obs: 0.16 / Rsym value: 0.16 / D res high: 1.7 Å / D res low: 19.858 Å / Num. obs: 8126 / % possible obs: 98.2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Diffraction reflection shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.17→19.86 Å / Num. obs: 24411 / % possible obs: 99.6 % / Observed criterion σ(I): 0 / 冗長度: 5.5 % / Biso Wilson estimate: 6.3 Å2 / Rmerge(I) obs: 0.094 / Rsym value: 0.094 / Net I/σ(I): 3.735 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | 解像度: 1.17→1.23 Å / 冗長度: 5.9 % / Rmerge(I) obs: 0.52 / Mean I/σ(I) obs: 1.3 / Rsym value: 0.52 / % possible all: 98.8 |
-位相決定
| 位相決定 | 手法:  単波長異常分散 単波長異常分散 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MAD set site |
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  単波長異常分散 / 解像度: 1.17→19.86 Å / Num. parameters: 6402 / Num. restraintsaints: 9922 / Occupancy max: 1 / Occupancy min: 0.22 / 交差検証法: FREE R / σ(F): 4 / 立体化学のターゲット値: ENGH AND HUBER 単波長異常分散 / 解像度: 1.17→19.86 Å / Num. parameters: 6402 / Num. restraintsaints: 9922 / Occupancy max: 1 / Occupancy min: 0.22 / 交差検証法: FREE R / σ(F): 4 / 立体化学のターゲット値: ENGH AND HUBER詳細: ANISOTROPIC REFINEMENT REDUCED FREE R (NO CUTOFF) BY 4%
| ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 10.739 Å2 | ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.17→19.86 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.17→1.22 Å /
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj