| 登録情報 | データベース: PDB / ID: 3ezi
|
|---|
| タイトル | Crystal Structure of the E. coli Histidine Kinase NarX Sensor Domain without Ligand |
|---|
 要素 要素 | Nitrate/nitrite sensor protein narX |
|---|
 キーワード キーワード | TRANSFERASE / histidine kinase sensor domain / Cell inner membrane / Cell membrane / Kinase / Membrane / Nitrate assimilation / Phosphoprotein / Transmembrane / Two-component regulatory system |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cellular response to nitrate / cellular response to nitrite / histidine kinase / phosphoprotein phosphatase activity / phosphorelay sensor kinase activity / nitrate assimilation / outer membrane-bounded periplasmic space / DNA damage response / signal transduction / protein homodimerization activity ...cellular response to nitrate / cellular response to nitrite / histidine kinase / phosphoprotein phosphatase activity / phosphorelay sensor kinase activity / nitrate assimilation / outer membrane-bounded periplasmic space / DNA damage response / signal transduction / protein homodimerization activity / ATP binding / plasma membrane類似検索 - 分子機能 Histidine kinase NarX, sensor domain / Signal transduction histidine kinase, nitrate/nitrite-sensing / NarX-like, N-terminal domain superfamily / NarX-like, N-terminal / Type IV pili methyl-accepting chemotaxis transducer N-term / Signal transduction histidine kinase, subgroup 3, dimerisation and phosphoacceptor domain / Histidine kinase / HAMP domain / HAMP (Histidine kinases, Adenylyl cyclases, Methyl binding proteins, Phosphatases) domain / HAMP domain profile. ...Histidine kinase NarX, sensor domain / Signal transduction histidine kinase, nitrate/nitrite-sensing / NarX-like, N-terminal domain superfamily / NarX-like, N-terminal / Type IV pili methyl-accepting chemotaxis transducer N-term / Signal transduction histidine kinase, subgroup 3, dimerisation and phosphoacceptor domain / Histidine kinase / HAMP domain / HAMP (Histidine kinases, Adenylyl cyclases, Methyl binding proteins, Phosphatases) domain / HAMP domain profile. / HAMP domain / Histidine kinase domain / Histidine kinase domain profile. / Four Helix Bundle (Hemerythrin (Met), subunit A) / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase / Histidine kinase/HSP90-like ATPase superfamily / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 ISOPROPYL ALCOHOL / Nitrate/nitrite sensor protein NarX類似検索 - 構成要素 |
|---|
| 生物種 |   Escherichia coli K12 (大腸菌) Escherichia coli K12 (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å |
|---|
 データ登録者 データ登録者 | Cheung, J. / Hendrickson, W.A. |
|---|
 引用 引用 |  ジャーナル: Structure / 年: 2009 ジャーナル: Structure / 年: 2009
タイトル: Structural Analysis of Ligand Stimulation of the Histidine Kinase NarX.
著者: Cheung, J. / Hendrickson, W.A. |
|---|
| 履歴 | | 登録 | 2008年10月22日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2008年12月23日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2017年10月25日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.3 | 2021年6月30日 | Group: Data collection / Derived calculations / カテゴリ: diffrn_detector / struct_site
Item: _diffrn_detector.detector / _struct_site.pdbx_auth_asym_id ..._diffrn_detector.detector / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.4 | 2023年12月27日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Structure / 年: 2009
ジャーナル: Structure / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3ezi.cif.gz
3ezi.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3ezi.ent.gz
pdb3ezi.ent.gz PDB形式
PDB形式 3ezi.json.gz
3ezi.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3ezi_validation.pdf.gz
3ezi_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3ezi_full_validation.pdf.gz
3ezi_full_validation.pdf.gz 3ezi_validation.xml.gz
3ezi_validation.xml.gz 3ezi_validation.cif.gz
3ezi_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ez/3ezi
https://data.pdbj.org/pub/pdb/validation_reports/ez/3ezi ftp://data.pdbj.org/pub/pdb/validation_reports/ez/3ezi
ftp://data.pdbj.org/pub/pdb/validation_reports/ez/3ezi リンク
リンク 集合体
集合体


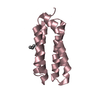

 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X4A / 波長: 0.9795 Å
/ ビームライン: X4A / 波長: 0.9795 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 1.7→19.79 Å / Rfactor Rfree error: 0.006 / FOM work R set: 0.832 / Data cutoff high absF: 391484.438 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0
分子置換 / 解像度: 1.7→19.79 Å / Rfactor Rfree error: 0.006 / FOM work R set: 0.832 / Data cutoff high absF: 391484.438 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0  ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj






