| 登録情報 | データベース: PDB / ID: 2yhh
|
|---|
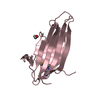
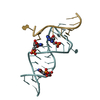
| タイトル | Microvirin:mannobiose complex |
|---|
 要素 要素 | MANNAN-BINDING LECTIN |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN / CYANOVIRIN FAMILY / CELL-CELL ATTACHMENT / ANTI-HIV PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
HIV-inactivating Protein, Cyanovirin-n / Cyanovirin-N / Cyanovirin-N / Cyanovirin-N superfamily / CVNH domain / CVNH / Roll / Mainly Beta類似検索 - ドメイン・相同性 2alpha-alpha-mannobiose / Mannan-binding lectin類似検索 - 構成要素 |
|---|
| 生物種 |  MICROCYSTIS AERUGINOSA (バクテリア) MICROCYSTIS AERUGINOSA (バクテリア) |
|---|
| 手法 | 溶液NMR / TORSION ANGLE, SIMULATED ANNEALING |
|---|
 データ登録者 データ登録者 | Hussan, S. / Bewley, C.A. / Clore, G.M. / Gustchina, E. / Ghirlando, R. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2011 ジャーナル: J.Biol.Chem. / 年: 2011
タイトル: Solution Structure of the Monovalent Lectin Microvirin in Complex with Man(Alpha)(1-2)Man Provides a Basis for Anti-HIV Activity with Low Toxicity.
著者: Shahzad-Ul-Hussan, S. / Gustchina, E. / Ghirlando, R. / Clore, G.M. / Bewley, C.A. |
|---|
| 履歴 | | 登録 | 2011年5月2日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2011年6月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年1月9日 | Group: Database references / Derived calculations ...Database references / Derived calculations / Other / Version format compliance |
|---|
| 改定 1.2 | 2019年7月3日 | Group: Data collection / Derived calculations / Other
カテゴリ: pdbx_database_proc / pdbx_database_status ...pdbx_database_proc / pdbx_database_status / pdbx_nmr_software / struct_conn
Item: _pdbx_database_status.date_of_cs_release / _pdbx_database_status.recvd_author_approval ..._pdbx_database_status.date_of_cs_release / _pdbx_database_status.recvd_author_approval / _pdbx_database_status.status_code_cs / _pdbx_nmr_software.name / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Other / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / entity_name_com / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_database_status / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_molecule_features / pdbx_nmr_spectrometer / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_validate_close_contact / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.auth_asym_id / _atom_site.auth_seq_id ..._atom_site.auth_asym_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.type / _pdbx_database_status.status_code_mr / _pdbx_nmr_spectrometer.model / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_validate_close_contact.auth_asym_id_1 / _pdbx_validate_close_contact.auth_asym_id_2 / _pdbx_validate_close_contact.auth_seq_id_1 / _pdbx_validate_close_contact.auth_seq_id_2 / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2024年11月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 MICROCYSTIS AERUGINOSA (バクテリア)
MICROCYSTIS AERUGINOSA (バクテリア) データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2011
ジャーナル: J.Biol.Chem. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2yhh.cif.gz
2yhh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2yhh.ent.gz
pdb2yhh.ent.gz PDB形式
PDB形式 2yhh.json.gz
2yhh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2yhh_validation.pdf.gz
2yhh_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2yhh_full_validation.pdf.gz
2yhh_full_validation.pdf.gz 2yhh_validation.xml.gz
2yhh_validation.xml.gz 2yhh_validation.cif.gz
2yhh_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/yh/2yhh
https://data.pdbj.org/pub/pdb/validation_reports/yh/2yhh ftp://data.pdbj.org/pub/pdb/validation_reports/yh/2yhh
ftp://data.pdbj.org/pub/pdb/validation_reports/yh/2yhh リンク
リンク 集合体
集合体
 要素
要素 MICROCYSTIS AERUGINOSA (バクテリア)
MICROCYSTIS AERUGINOSA (バクテリア)
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



 HSQC
HSQC