[English] 日本語
 Yorodumi
Yorodumi- PDB-2ww1: Structure of the Family GH92 Inverting Mannosidase BT3990 from Ba... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2ww1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Structure of the Family GH92 Inverting Mannosidase BT3990 from Bacteroides thetaiotaomicron VPI-5482 in complex with Thiomannobioside | |||||||||
 Components Components | PUTATIVE ALPHA-1,2-MANNOSIDASE | |||||||||
 Keywords Keywords | HYDROLASE / GLYCOSIDE HYDROLASE FAMILY 92 / GH92 / BT3990 | |||||||||
| Function / homology |  Function and homology information Function and homology informationpeptide-N4-(N-acetyl-beta-glucosaminyl)asparagine amidase activity / glycoprotein catabolic process / carbohydrate binding / carbohydrate metabolic process / metal ion binding / cytosol Similarity search - Function | |||||||||
| Biological species |  BACTEROIDES THETAIOTAOMICRON (bacteria) BACTEROIDES THETAIOTAOMICRON (bacteria) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.25 Å MOLECULAR REPLACEMENT / Resolution: 2.25 Å | |||||||||
 Authors Authors | Suits, M.D.L. / Zhu, Y. / Thompson, A. / Gilbert, H.J. / Davies, G.J. | |||||||||
 Citation Citation |  Journal: Nat.Chem.Biol. / Year: 2010 Journal: Nat.Chem.Biol. / Year: 2010Title: Mechanistic Insights Into a Ca2+-Dependent Family of A-Mannosidases in a Human Gut Symbiont. Authors: Zhu, Y. / Suits, M.D.L. / Thompson, A. / Chavan, S. / Dinev, Z. / Dumon, C. / Smith, N. / Moremen, K.W. / Xiang, Y. / Siriwardena, A. / Williams, S.J. / Gilbert, H.J. / Davies, G.J. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2ww1.cif.gz 2ww1.cif.gz | 604.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2ww1.ent.gz pdb2ww1.ent.gz | 498.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2ww1.json.gz 2ww1.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ww/2ww1 https://data.pdbj.org/pub/pdb/validation_reports/ww/2ww1 ftp://data.pdbj.org/pub/pdb/validation_reports/ww/2ww1 ftp://data.pdbj.org/pub/pdb/validation_reports/ww/2ww1 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2wvxSC  2wvyC  2wvzC  2ww0C  2ww2C  2ww3C  2wzsC  2wx8 C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments: Component-ID: 1 / Ens-ID: 1 / Beg auth comp-ID: TRP / Beg label comp-ID: TRP / End auth comp-ID: TYR / End label comp-ID: TYR / Refine code: 3 / Auth seq-ID: 22 - 750 / Label seq-ID: 3 - 731
NCS oper: (Code: given) |
- Components
Components
| #1: Protein | Mass: 85690.859 Da / Num. of mol.: 4 / Fragment: RESIDUES 20-755 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  BACTEROIDES THETAIOTAOMICRON (bacteria) BACTEROIDES THETAIOTAOMICRON (bacteria)Strain: VPI-5482 / Plasmid: PET 22 / Production host:  #2: Polysaccharide | alpha-D-mannopyranose-(1-2)-methyl 2-thio-alpha-D-mannopyranoside / methyl 2-S-alpha-D-mannopyranosyl-2-thio-alpha-D-mannopyranoside 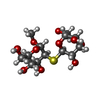  Type: oligosaccharide, Oligosaccharide / Class: Substrate analog / Mass: 372.389 Da / Num. of mol.: 4 Type: oligosaccharide, Oligosaccharide / Class: Substrate analog / Mass: 372.389 Da / Num. of mol.: 4Source method: isolated from a genetically manipulated source Details: oligosaccharide with S-glycosidic bond between monosaccharides References: methyl 2-S-alpha-D-mannopyranosyl-2-thio-alpha-D-mannopyranoside #3: Water | ChemComp-HOH / | Nonpolymer details | METHYL-2-S-(ALPHA-D-MANNOPYRAN | Sequence details | FIRST 19 RESIDUES WERE REMOVED DURING CLONING | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.75 Å3/Da / Density % sol: 55.4 % / Description: NONE |
|---|---|
| Crystal grow | Details: 20% 3350 PEG, 0.1 M (4-(2-HYDROXYETHYL)-1-PIPERAZINEETHANESULFONIC ACID) (PH 7.0), 0.15 M K2SO4 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID23-1 / Wavelength: 0.9762 / Beamline: ID23-1 / Wavelength: 0.9762 |
| Detector | Type: ADSC CCD / Detector: CCD / Date: Mar 17, 2009 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9762 Å / Relative weight: 1 |
| Reflection | Resolution: 2.25→44.6 Å / Num. obs: 174544 / % possible obs: 100 % / Observed criterion σ(I): 2 / Redundancy: 4.2 % / Biso Wilson estimate: 25.3 Å2 / Rmerge(I) obs: 0.14 / Net I/σ(I): 7.1 |
| Reflection shell | Resolution: 2.25→2.37 Å / Redundancy: 3.9 % / Rmerge(I) obs: 0.47 / Mean I/σ(I) obs: 3 / % possible all: 99.9 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 2WVX Resolution: 2.25→185.16 Å / Cor.coef. Fo:Fc: 0.932 / Cor.coef. Fo:Fc free: 0.906 / SU B: 11.86 / SU ML: 0.133 / TLS residual ADP flag: LIKELY RESIDUAL / Cross valid method: THROUGHOUT / ESU R: 0.27 / ESU R Free: 0.196 / Stereochemistry target values: MAXIMUM LIKELIHOOD Details: HYDROGENS HAVE BEEN ADDED IN THE ONLY. U VALUES RESIDUAL ONLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 13.722 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.25→185.16 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller




















 PDBj
PDBj


