| 登録情報 | データベース: PDB / ID: 2wbn
|
|---|
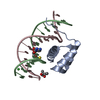
| タイトル | Crystal structure of the g2p (large terminase) nuclease domain from the bacteriophage SPP1 |
|---|
 要素 要素 | TERMINASE LARGE SUBUNIT |
|---|
 キーワード キーワード | VIRAL PROTEIN / LARGE TERMINASE / NUCLEASE / DNA PACKAGING |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
viral terminase, large subunit / viral DNA genome packaging / nuclease activity / chromosome organization / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / endonuclease activity / 加水分解酵素; エステル加水分解酵素 / ATP hydrolysis activity / ATP binding / metal ion binding類似検索 - 分子機能 Phage terminase large subunit, C-terminal / Terminase, large subunit SPP1-like / Nucleotidyltransferase; domain 5 - #280 / Terminase RNAseH like domain / Phage terminase large subunit, N-terminal / Phage terminase large subunit / Bacteriophage terminase, large subunit / Nucleotidyltransferase; domain 5 / P-loop containing nucleoside triphosphate hydrolase / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  BACILLUS PHAGE SPP1 (ファージ) BACILLUS PHAGE SPP1 (ファージ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.9 Å 多波長異常分散 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Smits, C. / Chechik, M. / Kovalevskiy, O.V. / Shevtsov, M.B. / Foster, A.W. / Alonso, J.C. / Antson, A.A. |
|---|
 引用 引用 |  ジャーナル: Embo Rep. / 年: 2009 ジャーナル: Embo Rep. / 年: 2009
タイトル: Structural Basis for the Nuclease Activity of a Bacteriophage Large Terminase.
著者: Smits, C. / Chechik, M. / Kovalevskiy, O.V. / Shevtsov, M.B. / Foster, A.W. / Alonso, J.C. / Antson, A.A. |
|---|
| 履歴 | | 登録 | 2009年3月2日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2009年3月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Advisory / Version format compliance |
|---|
| 改定 1.2 | 2011年10月12日 | Group: Database references / Derived calculations |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 BACILLUS PHAGE SPP1 (ファージ)
BACILLUS PHAGE SPP1 (ファージ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 1.9 Å
多波長異常分散 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Embo Rep. / 年: 2009
ジャーナル: Embo Rep. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2wbn.cif.gz
2wbn.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2wbn.ent.gz
pdb2wbn.ent.gz PDB形式
PDB形式 2wbn.json.gz
2wbn.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2wbn_validation.pdf.gz
2wbn_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2wbn_full_validation.pdf.gz
2wbn_full_validation.pdf.gz 2wbn_validation.xml.gz
2wbn_validation.xml.gz 2wbn_validation.cif.gz
2wbn_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/wb/2wbn
https://data.pdbj.org/pub/pdb/validation_reports/wb/2wbn ftp://data.pdbj.org/pub/pdb/validation_reports/wb/2wbn
ftp://data.pdbj.org/pub/pdb/validation_reports/wb/2wbn リンク
リンク 集合体
集合体
 要素
要素 BACILLUS PHAGE SPP1 (ファージ) / プラスミド: PYM20 / 発現宿主:
BACILLUS PHAGE SPP1 (ファージ) / プラスミド: PYM20 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: BM14 / 波長: 0.9184
/ ビームライン: BM14 / 波長: 0.9184  解析
解析 多波長異常分散
多波長異常分散 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj