+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2sn3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
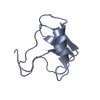
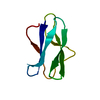
| タイトル | STRUCTURE OF SCORPION TOXIN VARIANT-3 AT 1.2 ANGSTROMS RESOLUTION | |||||||||
 要素 要素 | SCORPION NEUROTOXIN (VARIANT 3) | |||||||||
 キーワード キーワード | TOXIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sodium channel inhibitor activity / defense response / toxin activity / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / 解像度: 1.2 Å X線回折 / 解像度: 1.2 Å | |||||||||
 データ登録者 データ登録者 | Zhao, B. / Carson, M. / Ealick, S.E. / Bugg, C.E. | |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1992 ジャーナル: J.Mol.Biol. / 年: 1992タイトル: Structure of scorpion toxin variant-3 at 1.2 A resolution. 著者: Zhao, B. / Carson, M. / Ealick, S.E. / Bugg, C.E. #1:  ジャーナル: J.Mol.Biol. / 年: 1983 ジャーナル: J.Mol.Biol. / 年: 1983タイトル: Structure of Variant-3 Scorpion Neurotoxin from Centruroides Sculpturatus Ewing, Refined at 1.8 Angstroms Resolution 著者: Almassy, R.J. / Fontecilla-Camps, J.C. / Suddath, F.L. / Bugg, C.E. #2:  ジャーナル: Trends Biochem.Sci.(Pers. Ed.) / 年: 1981 ジャーナル: Trends Biochem.Sci.(Pers. Ed.) / 年: 1981タイトル: Architecture of Scorpion Neurotoxins. A Class of Membrane-Binding Proteins 著者: Fontecilla-Camps, J.C. / Almassy, R.J. / Ealick, S.E. / Suddath, F.L. / Watt, D.D. / Feldmann, R.J. / Bugg, C.E. #3:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1980 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1980タイトル: Three-Dimensional Structure of a Protein from Scorpion Venom. A New Structural Class of Neurotoxins 著者: Fontecilla-Camps, J.C. / Almassy, R.J. / Suddath, F.L. / Watt, D.D. / Bugg, C.E. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2sn3.cif.gz 2sn3.cif.gz | 36.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2sn3.ent.gz pdb2sn3.ent.gz | 26 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2sn3.json.gz 2sn3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2sn3_validation.pdf.gz 2sn3_validation.pdf.gz | 380.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2sn3_full_validation.pdf.gz 2sn3_full_validation.pdf.gz | 382 KB | 表示 | |
| XML形式データ |  2sn3_validation.xml.gz 2sn3_validation.xml.gz | 3.7 KB | 表示 | |
| CIF形式データ |  2sn3_validation.cif.gz 2sn3_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/sn/2sn3 https://data.pdbj.org/pub/pdb/validation_reports/sn/2sn3 ftp://data.pdbj.org/pub/pdb/validation_reports/sn/2sn3 ftp://data.pdbj.org/pub/pdb/validation_reports/sn/2sn3 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: CIS PROLINE - PRO 59 |
- 要素
要素
| #1: タンパク質 | 分子量: 7096.018 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  |
|---|---|
| #2: 化合物 | ChemComp-MPD / ( |
| #3: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.52 % | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 7.1 / 手法: 蒸気拡散法 | ||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 反射 | *PLUS Num. obs: 18909 / Observed criterion σ(I): 1 |
|---|
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.2→10 Å / σ(F): 0 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.2→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 1.2 Å / 最低解像度: 10 Å / Num. reflection all: 17706 / σ(F): 0 / Rfactor all: 0.192 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj




