| 登録情報 | データベース: PDB / ID: 2pnx
|
|---|
| タイトル | The PHD finger of ING4 in complex with an H3K4Me3 histone peptide |
|---|
 要素 要素 | - H3K4Me3 peptide
- Inhibitor of growth protein 4
|
|---|
 キーワード キーワード | GENE REGULATION / protein-peptide complex / chromatin / zinc finger / histone |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
DNA replication-dependent chromatin disassembly / regulation of DNA biosynthetic process / negative regulation of growth / intermediate filament cytoskeleton / histone H3K4me3 reader activity / regulation of cell cycle G2/M phase transition / positive regulation of DNA damage response, signal transduction by p53 class mediator / protein acetylation / : / histone acetyltransferase complex ...DNA replication-dependent chromatin disassembly / regulation of DNA biosynthetic process / negative regulation of growth / intermediate filament cytoskeleton / histone H3K4me3 reader activity / regulation of cell cycle G2/M phase transition / positive regulation of DNA damage response, signal transduction by p53 class mediator / protein acetylation / : / histone acetyltransferase complex / regulation of cell growth / HATs acetylate histones / transcription coactivator activity / regulation of cell cycle / positive regulation of apoptotic process / chromatin remodeling / negative regulation of cell population proliferation / negative regulation of DNA-templated transcription / apoptotic process / regulation of DNA-templated transcription / zinc ion binding / nucleoplasm / nucleus / cytosol類似検索 - 分子機能 Inhibitor of growth protein, N-terminal histone-binding / Inhibitor of growth proteins N-terminal histone-binding / Inhibitor of growth proteins N-terminal histone-binding / ING family / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-finger ...Inhibitor of growth protein, N-terminal histone-binding / Inhibitor of growth proteins N-terminal histone-binding / Inhibitor of growth proteins N-terminal histone-binding / ING family / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Zinc finger, PHD-type, conserved site / Zinc finger PHD-type signature. / Zinc finger PHD-type profile. / Zinc finger, PHD-finger / Zinc finger, PHD-type / PHD zinc finger / Zinc finger, FYVE/PHD-type / Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.8 Å 多波長異常分散 / 解像度: 1.8 Å |
|---|
 データ登録者 データ登録者 | Champagne, K.S. / Johnson, K. / Kutateladze, T.G. |
|---|
 引用 引用 |  ジャーナル: Mol.Cell / 年: 2009 ジャーナル: Mol.Cell / 年: 2009
タイトル: ING4 mediates crosstalk between histone H3 K4 trimethylation and H3 acetylation to attenuate cellular transformation
著者: Hung, T. / Binda, O. / Champagne, K.S. / Kuo, A.J. / Johnson, K. / Chang, H.Y. / Simon, M.D. / Kutateladze, T.G. / Gozani, O. |
|---|
| 履歴 | | 登録 | 2007年4月25日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2008年4月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2025年3月26日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_struct_conn_angle / struct_conn / struct_conn_type / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn_type.id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 / 解像度: 1.8 Å
多波長異常分散 / 解像度: 1.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Mol.Cell / 年: 2009
ジャーナル: Mol.Cell / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2pnx.cif.gz
2pnx.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2pnx.ent.gz
pdb2pnx.ent.gz PDB形式
PDB形式 2pnx.json.gz
2pnx.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2pnx_validation.pdf.gz
2pnx_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2pnx_full_validation.pdf.gz
2pnx_full_validation.pdf.gz 2pnx_validation.xml.gz
2pnx_validation.xml.gz 2pnx_validation.cif.gz
2pnx_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/pn/2pnx
https://data.pdbj.org/pub/pdb/validation_reports/pn/2pnx ftp://data.pdbj.org/pub/pdb/validation_reports/pn/2pnx
ftp://data.pdbj.org/pub/pdb/validation_reports/pn/2pnx リンク
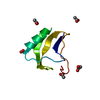
リンク 集合体
集合体

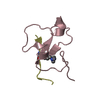
 要素
要素 Homo sapiens (ヒト) / 遺伝子: ING4 / プラスミド: pGEX-2T / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ING4 / プラスミド: pGEX-2T / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 4.2.2 / 波長: 1.282, 1.283, 1.257
/ ビームライン: 4.2.2 / 波長: 1.282, 1.283, 1.257 解析
解析 多波長異常分散 / 解像度: 1.8→34.08 Å / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber / 詳細: THE FRIEDEL PAIRS WERE USED FOR PHASING.
多波長異常分散 / 解像度: 1.8→34.08 Å / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber / 詳細: THE FRIEDEL PAIRS WERE USED FOR PHASING. ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj




