| 登録情報 | データベース: PDB / ID: 2p81
|
|---|
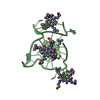
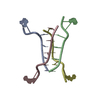
| タイトル | Engrailed homeodomain helix-turn-helix motif |
|---|
 要素 要素 | Segmentation polarity homeobox protein engrailed |
|---|
 キーワード キーワード | TRANSCRIPTION / helix-turn-helix motif / motif / engrailed / homeodomain / DNA binding / protein folding / intermediate / native |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
posterior compartment specification / analia development / anterior head segmentation / posterior head segmentation / anterior/posterior lineage restriction, imaginal disc / trunk segmentation / genital disc development / genital disc anterior/posterior pattern formation / spiracle morphogenesis, open tracheal system / wing disc anterior/posterior pattern formation ...posterior compartment specification / analia development / anterior head segmentation / posterior head segmentation / anterior/posterior lineage restriction, imaginal disc / trunk segmentation / genital disc development / genital disc anterior/posterior pattern formation / spiracle morphogenesis, open tracheal system / wing disc anterior/posterior pattern formation / wing disc morphogenesis / neuroblast fate determination / imaginal disc-derived wing vein specification / segment polarity determination / ventral midline development / compartment pattern specification / gonad development / axon guidance / RNA polymerase II transcription regulatory region sequence-specific DNA binding / DNA-binding transcription repressor activity, RNA polymerase II-specific / regulation of gene expression / sequence-specific DNA binding / negative regulation of neuron apoptotic process / DNA-binding transcription factor activity, RNA polymerase II-specific / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of gene expression / regulation of transcription by RNA polymerase II / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / nucleus類似検索 - 分子機能 Homeobox domain engrailed / Homeobox engrailed, C-terminal / Homeobox engrailed-type, conserved site / Engrailed homeobox C-terminal signature domain / 'Homeobox' engrailed-type protein signature. / : / Helix-turn-helix motif / Homeobox domain, metazoa / Homeobox, conserved site / 'Homeobox' domain signature. ...Homeobox domain engrailed / Homeobox engrailed, C-terminal / Homeobox engrailed-type, conserved site / Engrailed homeobox C-terminal signature domain / 'Homeobox' engrailed-type protein signature. / : / Helix-turn-helix motif / Homeobox domain, metazoa / Homeobox, conserved site / 'Homeobox' domain signature. / Homeodomain / 'Homeobox' domain profile. / Homeodomain / Homeobox domain / Homeobox-like domain superfamily類似検索 - ドメイン・相同性 Segmentation polarity homeobox protein engrailed類似検索 - 構成要素 |
|---|
| 生物種 |   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
|---|
| 手法 | 溶液NMR |
|---|
 データ登録者 データ登録者 | Religa, T.L. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007
タイトル: The helix-turn-helix motif as an ultrafast independently folding domain: The pathway of folding of Engrailed homeodomain.
著者: Religa, T.L. / Johnson, C.M. / Vu, D.M. / Brewer, S.H. / Dyer, R.B. / Fersht, A.R. |
|---|
| 履歴 | | 登録 | 2007年3月21日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2007年6月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2021年10月20日 | Group: Database references / Derived calculations
カテゴリ: database_2 / pdbx_struct_assembly ...database_2 / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. |
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007
ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2p81.cif.gz
2p81.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2p81.ent.gz
pdb2p81.ent.gz PDB形式
PDB形式 2p81.json.gz
2p81.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2p81_validation.pdf.gz
2p81_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2p81_full_validation.pdf.gz
2p81_full_validation.pdf.gz 2p81_validation.xml.gz
2p81_validation.xml.gz 2p81_validation.cif.gz
2p81_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/p8/2p81
https://data.pdbj.org/pub/pdb/validation_reports/p8/2p81 ftp://data.pdbj.org/pub/pdb/validation_reports/p8/2p81
ftp://data.pdbj.org/pub/pdb/validation_reports/p8/2p81 リンク
リンク 集合体
集合体
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
 NMRPipe
NMRPipe