+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2oru | ||||||
|---|---|---|---|---|---|---|---|
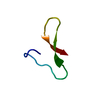
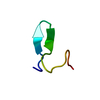
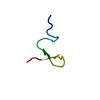
| タイトル | Solution structure of xtz1-peptide, a beta-hairpin peptide with a structured extension | ||||||
 要素 要素 | xtz1-peptide | ||||||
 キーワード キーワード | DE NOVO PROTEIN / hairpin | ||||||
| 手法 | 溶液NMR / Simulated annealing, TORSION ANGLE DYNAMICS | ||||||
 データ登録者 データ登録者 | Campbell, R.E. | ||||||
 引用 引用 |  ジャーナル: Chembiochem / 年: 2007 ジャーナル: Chembiochem / 年: 2007タイトル: In Vivo Screening Identifies a Highly Folded beta-Hairpin Peptide with a Structured Extension. 著者: Cheng, Z. / Miskolzie, M. / Campbell, R.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2oru.cif.gz 2oru.cif.gz | 117.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2oru.ent.gz pdb2oru.ent.gz | 84.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2oru.json.gz 2oru.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/or/2oru https://data.pdbj.org/pub/pdb/validation_reports/or/2oru ftp://data.pdbj.org/pub/pdb/validation_reports/or/2oru ftp://data.pdbj.org/pub/pdb/validation_reports/or/2oru | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 2551.854 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 2 mM xtz1-peptide; 90% H2O, 10% D2O, pH 6, 0.010 ml of 0.1% DSS 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 2 mM peptide / pH: 6 / 圧: AMBIENT / 温度: 288 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Varian INOVA / 製造業者: Varian / モデル: INOVA / 磁場強度: 600 MHz |
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: Simulated annealing, TORSION ANGLE DYNAMICS / ソフトェア番号: 1 詳細: Chemical shift assignments were manually done. Automated NOESY assignment and structure ensemble calculation was performed with CYANA v.2.1. A total of 431 unassigned NOESY peaks with ...詳細: Chemical shift assignments were manually done. Automated NOESY assignment and structure ensemble calculation was performed with CYANA v.2.1. A total of 431 unassigned NOESY peaks with intensity greater than ~1% of the most intense NOESY peak were input to CYANA. CYANA assigned 403 of 431 (93.5%) peaks, of which 309 were used as distance constraints in the structure calculation. | ||||||||||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
 CYANA
CYANA