+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2n3o | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of PTB RRM1(41-163) bound to an RNA stemloop containing a structured loop derived from viral internal ribosomal entry site RNA | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA BINDING PROTEIN/RNA / Polypyirimine tract binding protein / IRES / PTB / C-terminal helix formation / RNA BINDING PROTEIN-RNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of muscle cell differentiation / poly-pyrimidine tract binding / IRES-dependent viral translational initiation / positive regulation of calcineurin-NFAT signaling cascade / pre-mRNA binding / negative regulation of RNA splicing / FGFR2 alternative splicing / negative regulation of neuron differentiation / regulation of alternative mRNA splicing, via spliceosome / regulation of RNA splicing ...negative regulation of muscle cell differentiation / poly-pyrimidine tract binding / IRES-dependent viral translational initiation / positive regulation of calcineurin-NFAT signaling cascade / pre-mRNA binding / negative regulation of RNA splicing / FGFR2 alternative splicing / negative regulation of neuron differentiation / regulation of alternative mRNA splicing, via spliceosome / regulation of RNA splicing / negative regulation of mRNA splicing, via spliceosome / regulation of cell differentiation / Processing of Capped Intron-Containing Pre-mRNA / neurogenesis / mRNA Splicing - Major Pathway / RNA splicing / mRNA processing / mRNA binding / nucleolus / positive regulation of transcription by RNA polymerase II / RNA binding / extracellular exosome / nucleoplasm / nucleus / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||
| 手法 | 溶液NMR / simulated annealing, simulated annealing | ||||||
 データ登録者 データ登録者 | Maris, C. / Jayne, S.F. / Damberger, F.F. / Ravindranathan, S. / Allain, F.H.-T. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: C-terminal helix folding upon pyrimidine-rich hairpin binding to PTB RRM1. Implications for PTB function in Encephalomyocarditis virus IRES activity. 著者: Maris, C. / Jayne, S.F. / Damberger, F.F. / Ravindranathan, S. / Allain, F.H.-T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2n3o.cif.gz 2n3o.cif.gz | 1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2n3o.ent.gz pdb2n3o.ent.gz | 868 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2n3o.json.gz 2n3o.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2n3o_validation.pdf.gz 2n3o_validation.pdf.gz | 442.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2n3o_full_validation.pdf.gz 2n3o_full_validation.pdf.gz | 663.6 KB | 表示 | |
| XML形式データ |  2n3o_validation.xml.gz 2n3o_validation.xml.gz | 40.6 KB | 表示 | |
| CIF形式データ |  2n3o_validation.cif.gz 2n3o_validation.cif.gz | 75.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/n3/2n3o https://data.pdbj.org/pub/pdb/validation_reports/n3/2n3o ftp://data.pdbj.org/pub/pdb/validation_reports/n3/2n3o ftp://data.pdbj.org/pub/pdb/validation_reports/n3/2n3o | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ | |
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 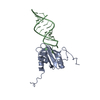
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 13375.167 Da / 分子数: 1 / 断片: UNP residues 41-163 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PTB, PTBP1 / プラスミド: pTYB11 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PTB, PTBP1 / プラスミド: pTYB11 / 発現宿主:  |
|---|---|
| #2: RNA鎖 | 分子量: 7308.343 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: Standard 3D NOESYs measured with 150 msec mixing time. 2D NOESY with 200 msec mixing time. NOESYs measured at 5C were obtained using 3919-watergate sequence before detection. RNA Constraints ...Text: Standard 3D NOESYs measured with 150 msec mixing time. 2D NOESY with 200 msec mixing time. NOESYs measured at 5C were obtained using 3919-watergate sequence before detection. RNA Constraints for all base-paired nucleotides except the closing G9-C15 basepair of the loop were derived from NOESY spectra obtained with free RNA - the NOE patterns were the same as for the RNA in the complex but signal to noise was better. 3D NOESYs of unlabeled RNA were measured with 200 msec mixing time, and 2D NOESY of unlabeled RNA was measured with 250 msec mixing time. |
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





























 HNCA
HNCA