+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2mm3 | ||||||
|---|---|---|---|---|---|---|---|
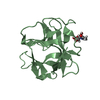
| タイトル | Solution NMR structure of the ternary complex of human ileal bile acid-binding protein with glycocholate and glycochenodeoxycholate | ||||||
 要素 要素 | Gastrotropin | ||||||
 キーワード キーワード | LIPID BINDING PROTEIN / lipid-binding protein / orthogonal beta sheets / positive binding cooperativity / site-selectivity / enterohepatic circulation | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / Triglyceride catabolism / fatty acid transport / Recycling of bile acids and salts / fatty acid binding / lipid metabolic process / negative regulation of cell population proliferation / lipid binding / nucleus / membrane ...NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / Triglyceride catabolism / fatty acid transport / Recycling of bile acids and salts / fatty acid binding / lipid metabolic process / negative regulation of cell population proliferation / lipid binding / nucleus / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / simulated annealing, torsion angle dynamics | ||||||
| Model details | closest to the average, model9 | ||||||
 データ登録者 データ登録者 | Horvath, G. / Egyed, O. / Bencsura, A. / Simon, A. / Tochtrop, G.P. / DeKoster, G.T. / Covey, D.F. / Cistola, D.P. / Toke, O. | ||||||
 引用 引用 |  ジャーナル: FEBS J. / 年: 2016 ジャーナル: FEBS J. / 年: 2016タイトル: Structural determinants of ligand binding in the ternary complex of human ileal bile acid binding protein with glycocholate and glycochenodeoxycholate obtained from solution NMR 著者: Horvath, G. / Bencsura, A. / Simon, A. / Tochtrop, G.P. / DeKoster, G.T. / Covey, D.F. / Cistola, D.P. / Toke, O. #1: ジャーナル: Biochemistry / 年: 2006 タイトル: Determinants of cooperativity and site selectivity in human ileal bile acid binding protein. 著者: Toke, O. / Monsey, J.D. / DeKoster, G.T. / Tochtrop, G.P. / Tang, C. / Cistola, D.P. #2: ジャーナル: J.Am.Chem.Soc. / 年: 2004 タイトル: A single hydroxyl group governs ligand site selectivity in human ileal bile acid binding protein. 著者: Tochtrop, G.P. / DeKoster, G.T. / Covey, D.F. / Cistola, D.P. #3: ジャーナル: Biochemistry / 年: 2007 タイトル: Kinetic mechanism of ligand binding in human ileal bile acid binding protein as determined by stopped-flow fluorescence analysis. 著者: Toke, O. / Monsey, J.D. / Cistola, D.P. #4: ジャーナル: Biochemistry / 年: 2012 タイトル: Internal motions and exchange processes in human ileal bile acid binding protein as studied by backbone (15)N nuclear magnetic resonance spectroscopy. 著者: Horvath, G. / Kiraly, P. / Tarkanyi, G. / Toke, O. #5:  ジャーナル: Biochemistry / 年: 2012 ジャーナル: Biochemistry / 年: 2012タイトル: Internal motions and exchange processes in human ileal bile acid binding protein as studied by backbone (15)N nuclear magnetic resonance spectroscopy 著者: Horvath, G. / Kiraly, P. / Tarkanyi, G. / Toke, O. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2mm3.cif.gz 2mm3.cif.gz | 457.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2mm3.ent.gz pdb2mm3.ent.gz | 390.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2mm3.json.gz 2mm3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2mm3_validation.pdf.gz 2mm3_validation.pdf.gz | 666.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2mm3_full_validation.pdf.gz 2mm3_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  2mm3_validation.xml.gz 2mm3_validation.xml.gz | 131.4 KB | 表示 | |
| CIF形式データ |  2mm3_validation.cif.gz 2mm3_validation.cif.gz | 94.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mm/2mm3 https://data.pdbj.org/pub/pdb/validation_reports/mm/2mm3 ftp://data.pdbj.org/pub/pdb/validation_reports/mm/2mm3 ftp://data.pdbj.org/pub/pdb/validation_reports/mm/2mm3 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14258.038 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: FABP6 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: FABP6 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-GCH / |
| #3: 化合物 | ChemComp-CHO / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 試料状態 | イオン強度: 70 / pH: 6.3 / 圧: ambient / 温度: 293 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian Varian NMR System / 製造業者: Varian / モデル: Varian NMR System / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing, torsion angle dynamics / ソフトェア番号: 1 | ||||||||||||||||||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 / 代表コンフォーマー: 9 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






 HSQC
HSQC