| 登録情報 | データベース: PDB / ID: 2mhq
|
|---|
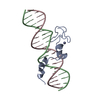
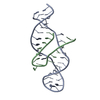
| タイトル | Solution structure of the major factor VIII binding region on von Willebrand factor |
|---|
 要素 要素 | von Willebrand factor |
|---|
 キーワード キーワード | BLOOD CLOTTING / von Willebrand factor / factor VIII / ensemble refinement |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Defective VWF binding to collagen type I / Enhanced cleavage of VWF variant by ADAMTS13 / Defective VWF cleavage by ADAMTS13 variant / Defective F8 binding to von Willebrand factor / Enhanced binding of GP1BA variant to VWF multimer:collagen / Defective binding of VWF variant to GPIb:IX:V / Weibel-Palade body / hemostasis / platelet alpha granule / Platelet Adhesion to exposed collagen ...Defective VWF binding to collagen type I / Enhanced cleavage of VWF variant by ADAMTS13 / Defective VWF cleavage by ADAMTS13 variant / Defective F8 binding to von Willebrand factor / Enhanced binding of GP1BA variant to VWF multimer:collagen / Defective binding of VWF variant to GPIb:IX:V / Weibel-Palade body / hemostasis / platelet alpha granule / Platelet Adhesion to exposed collagen / GP1b-IX-V activation signalling / p130Cas linkage to MAPK signaling for integrins / cell-substrate adhesion / Defective F8 cleavage by thrombin / Platelet Aggregation (Plug Formation) / GRB2:SOS provides linkage to MAPK signaling for Integrins / positive regulation of intracellular signal transduction / immunoglobulin binding / Integrin cell surface interactions / collagen binding / Intrinsic Pathway of Fibrin Clot Formation / Integrin signaling / extracellular matrix / platelet alpha granule lumen / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / platelet activation / response to wounding / integrin binding / blood coagulation / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / Platelet degranulation / protein-folding chaperone binding / : / protease binding / cell adhesion / endoplasmic reticulum / extracellular space / extracellular exosome / extracellular region / identical protein binding類似検索 - 分子機能 von Willebrand factor, VWA N-terminal domain / Von Willebrand factor / VWA N-terminal / Von Willebrand factor-like domain / : / C8 domain / Uncharacterised domain, cysteine-rich / C8 / von Willebrand factor, type D domain / von Willebrand factor type D domain ...von Willebrand factor, VWA N-terminal domain / Von Willebrand factor / VWA N-terminal / Von Willebrand factor-like domain / : / C8 domain / Uncharacterised domain, cysteine-rich / C8 / von Willebrand factor, type D domain / von Willebrand factor type D domain / VWFD domain profile. / von Willebrand factor (vWF) type D domain / von Willebrand factor (vWF) type C domain / Trypsin Inhibitor-like, cysteine rich domain / Serine protease inhibitor-like superfamily / Trypsin Inhibitor like cysteine rich domain / von Willebrand factor type C domain / C-terminal cystine knot signature. / C-terminal cystine knot domain profile. / Cystine knot, C-terminal / C-terminal cystine knot-like domain (CTCK) / VWFC domain signature. / VWFC domain profile. / von Willebrand factor (vWF) type C domain / VWFC domain / von Willebrand factor type A domain / VWFA domain profile. / von Willebrand factor (vWF) type A domain / von Willebrand factor, type A / von Willebrand factor A-like domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / torsion angle dynamics |
|---|
| Model details | Not applicable, model1 |
|---|
 データ登録者 データ登録者 | Shiltagh, N. / Kirkpatrick, J. / Cabrita, L.D. / McKinnon, T.A.J. / Thalassinos, K. / Tuddenham, E.G.D. / Hansen, D.F. |
|---|
 引用 引用 |  ジャーナル: Blood / 年: 2014 ジャーナル: Blood / 年: 2014
タイトル: Solution structure of the major factor VIII binding region on von Willebrand factor.
著者: Shiltagh, N. / Kirkpatrick, J. / Cabrita, L.D. / McKinnon, T.A. / Thalassinos, K. / Tuddenham, E.G. / Hansen, D.F. |
|---|
| 履歴 | | 登録 | 2013年12月2日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2014年5月14日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年8月24日 | Group: Data collection / Database references
カテゴリ: citation / citation_author ...citation / citation_author / database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation_author.name / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Other / カテゴリ: pdbx_database_status / Item: _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.3 | 2024年10月30日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI / _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Blood / 年: 2014
ジャーナル: Blood / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2mhq.cif.gz
2mhq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2mhq.ent.gz
pdb2mhq.ent.gz PDB形式
PDB形式 2mhq.json.gz
2mhq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2mhq_validation.pdf.gz
2mhq_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2mhq_full_validation.pdf.gz
2mhq_full_validation.pdf.gz 2mhq_validation.xml.gz
2mhq_validation.xml.gz 2mhq_validation.cif.gz
2mhq_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mh/2mhq
https://data.pdbj.org/pub/pdb/validation_reports/mh/2mhq ftp://data.pdbj.org/pub/pdb/validation_reports/mh/2mhq
ftp://data.pdbj.org/pub/pdb/validation_reports/mh/2mhq リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: VWF, F8VWF / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: VWF, F8VWF / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj













 HNCA
HNCA