| 登録情報 | データベース: PDB / ID: 2la5
|
|---|
| タイトル | RNA Duplex-Quadruplex Junction Complex with FMRP RGG peptide |
|---|
 要素 要素 | - Fragile X mental retardation 1 protein
- RNA (36-MER)
|
|---|
 キーワード キーワード | RNA Binding Protein/RNA / RNA Binding Protein-RNA complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of intracellular transport of viral material / regulation of translation at presynapse, modulating synaptic transmission / dendritic filopodium / host-mediated perturbation of viral RNA genome replication / poly(G) binding / histone H3 reader activity / positive regulation of miRNA-mediated gene silencing / regulation of neuronal action potential / negative regulation of miRNA-mediated gene silencing / neuronal ribonucleoprotein granule ...positive regulation of intracellular transport of viral material / regulation of translation at presynapse, modulating synaptic transmission / dendritic filopodium / host-mediated perturbation of viral RNA genome replication / poly(G) binding / histone H3 reader activity / positive regulation of miRNA-mediated gene silencing / regulation of neuronal action potential / negative regulation of miRNA-mediated gene silencing / neuronal ribonucleoprotein granule / animal organ development / negative regulation of voltage-gated calcium channel activity / negative regulation of long-term synaptic depression / regulation of dendritic spine development / RNA strand annealing activity / chromocenter / filopodium tip / positive regulation of long-term neuronal synaptic plasticity / regulation of filopodium assembly / regulation of neurotransmitter secretion / negative regulation of synaptic vesicle exocytosis / N6-methyladenosine-containing RNA reader activity / poly(A) binding / siRNA binding / positive regulation of proteasomal protein catabolic process / membraneless organelle assembly / growth cone filopodium / regulatory ncRNA-mediated gene silencing / sequence-specific mRNA binding / miRNA binding / positive regulation of filopodium assembly / poly(U) RNA binding / glutamate receptor signaling pathway / intracellular membraneless organelle / regulation of alternative mRNA splicing, via spliceosome / positive regulation of dendritic spine development / dynein complex binding / positive regulation of receptor internalization / chromosome, centromeric region / glial cell projection / mRNA transport / mRNA export from nucleus / Cajal body / negative regulation of cytoplasmic translation / translation regulator activity / translation initiation factor binding / translation repressor activity / axon terminus / signaling adaptor activity / negative regulation of translational initiation / regulation of mRNA stability / stress granule assembly / RNA splicing / positive regulation of translation / mRNA 3'-UTR binding / cell projection / molecular condensate scaffold activity / mRNA 5'-UTR binding / cellular response to virus / RNA stem-loop binding / cytoplasmic ribonucleoprotein granule / mRNA processing / cytoplasmic stress granule / nervous system development / presynapse / chromosome / ribosome binding / growth cone / presynaptic membrane / G-quadruplex RNA binding / perikaryon / microtubule binding / dendritic spine / transmembrane transporter binding / postsynaptic membrane / neuron projection / postsynapse / negative regulation of translation / postsynaptic density / protein heterodimerization activity / ribonucleoprotein complex / axon / DNA repair / mRNA binding / dendrite / synapse / chromatin binding / nucleolus / perinuclear region of cytoplasm / protein homodimerization activity / RNA binding / nucleoplasm / identical protein binding / nucleus / membrane / cytosol / cytoplasm類似検索 - 分子機能 Fragile X messenger ribonucleoprotein 1, C-terminal region 2 / : / : / : / : / Fragile X messenger ribonucleoprotein 1, C-terminal region 2 / Fragile X messenger ribonucleoprotein 1, C-terminal core / Fragile X messenger ribonucleoprotein 1 / Synaptic functional regulator FMRP, KH0 domain / FMR1, tudor domain ...Fragile X messenger ribonucleoprotein 1, C-terminal region 2 / : / : / : / : / Fragile X messenger ribonucleoprotein 1, C-terminal region 2 / Fragile X messenger ribonucleoprotein 1, C-terminal core / Fragile X messenger ribonucleoprotein 1 / Synaptic functional regulator FMRP, KH0 domain / FMR1, tudor domain / Fragile X-related 1 protein core C terminal / FMRP KH0 domain / Fragile X messenger ribonucleoprotein 1, Tudor domain / Agenet-like domain profile. / Agenet-like domain / Agenet domain / KH domain / K Homology domain, type 1 / Type-1 KH domain profile. / K Homology domain, type 1 superfamily / K Homology domain / K homology RNA-binding domain類似検索 - ドメイン・相同性 RNA / RNA (> 10) / Fragile X messenger ribonucleoprotein 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / torsion angle dynamics, molecular dynamics |
|---|
| Model details | lowest energy, model 1 |
|---|
 データ登録者 データ登録者 | Phan, A. / Kuryavyi, V. / Darnell, J. / Serganov, A. / Majumdar, A. / Ilin, S. / Darnell, R. / Patel, D. |
|---|
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011
タイトル: Structure-function studies of FMRP RGG peptide recognition of an RNA duplex-quadruplex junction.
著者: Phan, A.T. / Kuryavyi, V. / Darnell, J.C. / Serganov, A. / Majumdar, A. / Ilin, S. / Raslin, T. / Polonskaia, A. / Chen, C. / Clain, D. / Darnell, R.B. / Patel, D.J. |
|---|
| 履歴 | | 登録 | 2011年3月3日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2011年6月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月20日 | Group: Database references |
|---|
| 改定 1.3 | 2024年5月1日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011
ジャーナル: Nat.Struct.Mol.Biol. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2la5.cif.gz
2la5.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2la5.ent.gz
pdb2la5.ent.gz PDB形式
PDB形式 2la5.json.gz
2la5.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2la5_validation.pdf.gz
2la5_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2la5_full_validation.pdf.gz
2la5_full_validation.pdf.gz 2la5_validation.xml.gz
2la5_validation.xml.gz 2la5_validation.cif.gz
2la5_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/la/2la5
https://data.pdbj.org/pub/pdb/validation_reports/la/2la5 ftp://data.pdbj.org/pub/pdb/validation_reports/la/2la5
ftp://data.pdbj.org/pub/pdb/validation_reports/la/2la5 リンク
リンク 集合体
集合体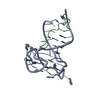
 要素
要素 Homo sapiens (ヒト) / 遺伝子: FMR1 / 参照: UniProt: Q06787
Homo sapiens (ヒト) / 遺伝子: FMR1 / 参照: UniProt: Q06787 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー




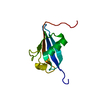








 PDBj
PDBj






























 HSQC
HSQC