+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2kiz | ||||||
|---|---|---|---|---|---|---|---|
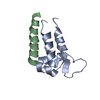
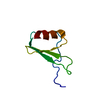
| タイトル | Solution structure of Arkadia RING-H2 finger domain | ||||||
 要素 要素 | E3 ubiquitin-protein ligase Arkadia | ||||||
 キーワード キーワード | METAL BINDING PROTEIN / ARKADIA / RING-H2 FINGER / E3 LIGASE / ZN BINDING DOMAIN / Metal-binding / Zinc-finger | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SUMO polymer binding / global genome nucleotide-excision repair / pattern specification process / positive regulation of transforming growth factor beta receptor signaling pathway / SMAD binding / positive regulation of protein ubiquitination / Downregulation of SMAD2/3:SMAD4 transcriptional activity / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / RING-type E3 ubiquitin transferase / PML body ...SUMO polymer binding / global genome nucleotide-excision repair / pattern specification process / positive regulation of transforming growth factor beta receptor signaling pathway / SMAD binding / positive regulation of protein ubiquitination / Downregulation of SMAD2/3:SMAD4 transcriptional activity / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / RING-type E3 ubiquitin transferase / PML body / Formation of Incision Complex in GG-NER / protein polyubiquitination / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / protein ubiquitination / protein-containing complex / zinc ion binding / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / torsion angle dynamics | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Kandias, N.G. / Chasapis, C.T. / Bentrop, D. / Episkopou, V. / Spyroulias, G.A. | ||||||
 引用 引用 |  ジャーナル: Proteins / 年: 2012 ジャーナル: Proteins / 年: 2012タイトル: NMR-based insights into the conformational and interaction properties of Arkadia RING-H2 E3 Ub ligase. 著者: Chasapis, C.T. / Kandias, N.G. / Episkopou, V. / Bentrop, D. / Spyroulias, G.A. #1: ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2009 タイトル: High yield expression and NMR characterization of Arkadia E3 ubiquitin ligase RING-H2 finger domain. 著者: Kandias, N.G. / Chasapis, C.T. / Bentrop, D. / Episkopou, V. / Spyroulias, G.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2kiz.cif.gz 2kiz.cif.gz | 732.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2kiz.ent.gz pdb2kiz.ent.gz | 623.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2kiz.json.gz 2kiz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ki/2kiz https://data.pdbj.org/pub/pdb/validation_reports/ki/2kiz ftp://data.pdbj.org/pub/pdb/validation_reports/ki/2kiz ftp://data.pdbj.org/pub/pdb/validation_reports/ki/2kiz | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 7960.034 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: RNF111 / プラスミド: pGEX / 発現宿主: Homo sapiens (ヒト) / 遺伝子: RNF111 / プラスミド: pGEX / 発現宿主:  | ||
|---|---|---|---|
| #2: 化合物 | | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法: 溶液NMR / 詳細: Recombinant RING finger polypeptide (RING-H2 type) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 1.0 mM [U-98% 13C; U-98% 15N] RING Finger monomer, 50 mM Pi, 90% H2O/10% D2O 溶媒系: 90% H2O/10% D2O | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||
| 試料状態 | イオン強度: 50 / pH: 6.9 / 圧: ambient / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker Avance / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics / ソフトェア番号: 1 | ||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 31 / 登録したコンフォーマーの数: 31 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj






 HSQC
HSQC