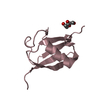
登録情報 データベース : PDB / ID : 2jmcタイトル Chimer between Spc-SH3 and P41 Spectrin alpha chain, brain and P41 peptide chimera キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Gallus gallus (ニワトリ)手法 / Model details Chimer between Spc-SH3 and decapeptide P41 starting from circular permutant S19P20s データ登録者 van Nuland, N.A.J. / Candel, A.M. / Martinez, J.C. / Conejero-Lara, F. / Bruix, M. ジャーナル : Febs Lett. / 年 : 2007タイトル : The high-resolution NMR structure of a single-chain chimeric protein mimicking a SH3-peptide complex著者 : Candel, A.M. / Conejero-Lara, F. / Martinez, J.C. / van Nuland, N.A.J. / Bruix, M. 履歴 登録 2006年11月2日 登録サイト / 処理サイト 改定 1.0 2007年4月24日 Provider / タイプ 改定 1.1 2008年5月1日 Group 改定 1.2 2011年7月13日 Group 改定 1.3 2022年3月9日 Group / Database references / Derived calculationsカテゴリ database_2 / pdbx_nmr_software ... database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details 改定 1.4 2023年12月20日 Group / Otherカテゴリ / chem_comp_bond / pdbx_database_statusItem 改定 1.5 2024年11月20日 Group / カテゴリ / pdbx_entry_details
すべて表示 表示を減らす Remark 999 SEQUENCE The author states that the chimera is based on a circular permutant of alpha-spectrin ... SEQUENCE The author states that the chimera is based on a circular permutant of alpha-spectrin SH3, named S19P20s from PDB entry 1TUC. The original N- and C-terminus of alpha-spectrin are connected and new N- and C-termini are introduced. Residues 5-47 correspond to residues 20-62 in PDB entry 1TUC. Residues 50-63 correspond to residues 4-17 in PDB entry 1TUC. S64 corresponds to residue 19 from PDB entry 1TUC. Residues 68-77 correspond to P41 peptide, see PDB entry 2JMA.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: Febs Lett. / 年: 2007
ジャーナル: Febs Lett. / 年: 2007 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2jmc.cif.gz
2jmc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2jmc.ent.gz
pdb2jmc.ent.gz PDB形式
PDB形式 2jmc.json.gz
2jmc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2jmc_validation.pdf.gz
2jmc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2jmc_full_validation.pdf.gz
2jmc_full_validation.pdf.gz 2jmc_validation.xml.gz
2jmc_validation.xml.gz 2jmc_validation.cif.gz
2jmc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/jm/2jmc
https://data.pdbj.org/pub/pdb/validation_reports/jm/2jmc ftp://data.pdbj.org/pub/pdb/validation_reports/jm/2jmc
ftp://data.pdbj.org/pub/pdb/validation_reports/jm/2jmc リンク
リンク 集合体
集合体
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj HNCA
HNCA