[English] 日本語
 Yorodumi
Yorodumi- PDB-2hmz: THE STRUCTURES OF MET AND AZIDOMET HEMERYTHRIN AT 1.66 ANGSTROMS ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2hmz | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | THE STRUCTURES OF MET AND AZIDOMET HEMERYTHRIN AT 1.66 ANGSTROMS RESOLUTION | |||||||||
 Components Components | HEMERYTHRIN | |||||||||
 Keywords Keywords | OXYGEN TRANSPORT | |||||||||
| Function / homology |  Function and homology information Function and homology information | |||||||||
| Biological species |  Themiste dyscritum (invertebrata) Themiste dyscritum (invertebrata) | |||||||||
| Method |  X-RAY DIFFRACTION / Resolution: 1.66 Å X-RAY DIFFRACTION / Resolution: 1.66 Å | |||||||||
 Authors Authors | Holmes, M.A. / Stenkamp, R.E. | |||||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 1991 Journal: J.Mol.Biol. / Year: 1991Title: Structures of met and azidomet hemerythrin at 1.66 A resolution. Authors: Holmes, M.A. / Stenkamp, R.E. #1:  Journal: Proc.Natl.Acad.Sci.USA / Year: 1985 Journal: Proc.Natl.Acad.Sci.USA / Year: 1985Title: Active Site Structures of Deoxyhemerythrin and Oxyhemerythrin Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. / Mccallum, J.D. / Sanders-Loehr, L. #2:  Journal: J.Am.Chem.Soc. / Year: 1984 Journal: J.Am.Chem.Soc. / Year: 1984Title: Binuclear Iron Complexes in Methemerythrin and Azidomethemerythrin at 2.0-Angstroms Resolution Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. #3:  Journal: Acta Crystallogr.,Sect.B / Year: 1983 Journal: Acta Crystallogr.,Sect.B / Year: 1983Title: Adjustment of Restraints in the Refinement of Methemerythrin and Azidomethemerythrin at 2.0 Angstroms Resolution Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. #4:  Journal: Acta Crystallogr.,Sect.B / Year: 1982 Journal: Acta Crystallogr.,Sect.B / Year: 1982Title: Restrained Least-Squares Refinement of Themiste Dyscritum Methydroxohemerythrin at 2.0 Angstroms Resolution Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. #5:  Journal: Nature / Year: 1981 Journal: Nature / Year: 1981Title: Structure of the Binuclear Iron Complex in Metazidohaemerythrin from Themiste Dyscritum at 2.2 Angstroms Resolution Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. / Sanders-Loehr, J. #6:  Journal: J.Mol.Biol. / Year: 1978 Journal: J.Mol.Biol. / Year: 1978Title: Crystallographic Studies of Azide, Thiocyanate and Perchlorate Complexes of Methemerythrin Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. #7:  Journal: Biochemistry / Year: 1978 Journal: Biochemistry / Year: 1978Title: Structure of Methemerythrin at 2.8-Angstroms Resolution. Computer Graphics Fit of an Averaged Electron Density Map Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. / Mcqueenjunior, J.E. #8:  Journal: J.Biol.Chem. / Year: 1978 Journal: J.Biol.Chem. / Year: 1978Title: Amino Acid Sequence of Hemerythrin from Themiste Dyscritum Authors: Loehr, J.S. / Lammers, P.J. / Brimhall, B. / Hermodson, M.A. #9:  Journal: Proc.Natl.Acad.Sci.USA / Year: 1976 Journal: Proc.Natl.Acad.Sci.USA / Year: 1976Title: Structure of the Iron Complex in Methemerythrin Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. #10:  Journal: J.Mol.Biol. / Year: 1976 Journal: J.Mol.Biol. / Year: 1976Title: Structure of Methemerythrin at 5 Angstroms Resolution Authors: Stenkamp, R.E. / Sieker, L.C. / Jensen, L.H. / Loehr, J.S. #11:  Journal: J.Mol.Biol. / Year: 1975 Journal: J.Mol.Biol. / Year: 1975Title: An X-Ray Crystallographic Study of Hemerythrin Authors: Loehr, J.S. / Meyerhoff, K.N. / Sieker, L.C. / Jensen, L.H. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2hmz.cif.gz 2hmz.cif.gz | 113.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2hmz.ent.gz pdb2hmz.ent.gz | 88.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2hmz.json.gz 2hmz.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/hm/2hmz https://data.pdbj.org/pub/pdb/validation_reports/hm/2hmz ftp://data.pdbj.org/pub/pdb/validation_reports/hm/2hmz ftp://data.pdbj.org/pub/pdb/validation_reports/hm/2hmz | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 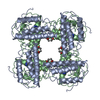
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| Unit cell |
| ||||||||
| Atom site foot note | 1: PRO 7 IS A CIS-PROLINE. 2: VAL 21 IS MODELED AS VAL AND ILE EACH WITH HALF OCCUPANCY. 3: ALA 64 IS MODELED AS AN ACETYLATED SERINE. 4: THE SIDE CHAINS OF THE FOLLOWING RESIDUES ARE DISORDERED: CHAIN A - 53, 72, 108 CHAIN B - 28, 53 CHAIN C - 60, 72 5: HYDROGEN BONDING WOULD ARGUE FOR INTERCHANGING THE SIDE-CHAIN N AND O ATOMS FOR RESIDUES ASN 40 AND GLU 59. |
- Components
Components
| #1: Protein | Mass: 13352.996 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Themiste dyscritum (invertebrata) / References: UniProt: P02246 Themiste dyscritum (invertebrata) / References: UniProt: P02246#2: Chemical | ChemComp-ACT / #3: Chemical | ChemComp-FEA / #4: Water | ChemComp-HOH / | Nonpolymer details | THE BINUCLEAR IRON COMPLEX (DESIGNATED FEA BELOW) CONSISTS OF TWO FE ATOMS BRIDGED BY A MU-OXYGEN ...THE BINUCLEAR IRON COMPLEX (DESIGNATED | Sequence details | RESIDUE 21 HAS BEEN MODELED BOTH AS VAL AND AS ILE. IT IS PRESENTED IN THE ATOM RECORDS BELOW AS ...RESIDUE 21 HAS BEEN MODELED BOTH AS VAL AND AS ILE. IT IS PRESENTED IN THE ATOM RECORDS BELOW AS ILE 21A FOLLOWED BY VAL 21B. ALL ATOMS OF THIS RESIDUE HAVE BEEN ASSIGNED OCCUPANCIE | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION X-RAY DIFFRACTION |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.84 Å3/Da / Density % sol: 56.67 % |
|---|---|
| Crystal grow | *PLUS Method: microdialysisDetails: referred to 'Loehr, J.S.', (1975) J.Mol.Biol., 91, 521-525 |
| Components of the solutions | *PLUS Conc.: 35 % / Common name: MPD |
- Processing
Processing
| Software | Name: PROFFT / Classification: refinement | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 1.66→10 Å Details: THE SIDE CHAINS OF THE FOLLOWING RESIDUES ARE DISORDERED: CHAIN A - 53, 72, 108 CHAIN B - 28, 53 CHAIN C - 60, 72
| ||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.66→10 Å
| ||||||||||||
| Refinement | *PLUS Highest resolution: 1.66 Å / Lowest resolution: 10 Å / Rfactor obs: 0.176 / Num. reflection obs: 68747 | ||||||||||||
| Solvent computation | *PLUS | ||||||||||||
| Displacement parameters | *PLUS | ||||||||||||
| Refine LS restraints | *PLUS
|
 Movie
Movie Controller
Controller









 PDBj
PDBj