| 登録情報 | データベース: PDB / ID: 2hem
|
|---|
| タイトル | NMR structure and Mg2+ binding of an RNA segment that underlies the L7/L12 stalk in the E.coli 50S ribosomal subunit. |
|---|
 要素 要素 | 5'-R(P*GP*GP*GP*AP*AP*GP*GP*CP*GP*CP*UP*UP*CP*GP*GP*CP*GP*UP*CP*GP*GP*CP*CP*C)-3'  キーワード キーワード | RNA / 23S rRNA / backbone dynamics / cis Watson-Crick G A pair / G A mismatch / helix 42 / L7/L12 stalk / Mg2+ binding / tandem (G A)2 |
|---|
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 | synthetic construct (人工物) |
|---|
| 手法 | 溶液NMR / distance geometry simulated annealing |
|---|
 データ登録者 データ登録者 | Zhao, Q. / Nagaswamy, U. / Lee, H. / Xia, Y. / Gao, X. / Fox, G. |
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2005 ジャーナル: Nucleic Acids Res. / 年: 2005
タイトル: NMR structure and Mg2+ binding of an RNA segment that underlies the L7/L12 stalk in the E.coli 50S ribosomal subunit
著者: Zhao, Q. / Nagaswamy, U. / Lee, H. / Xia, Y. / Huan, H.-C. / Gao, X. / Fox, G.E. |
|---|
| 履歴 | | 登録 | 2006年6月21日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2006年9月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年5月1日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2020年6月24日 | Group: Database references / Derived calculations / Source and taxonomy
カテゴリ: ndb_struct_na_base_pair / pdbx_entity_src_syn ...ndb_struct_na_base_pair / pdbx_entity_src_syn / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref / struct_ref_seq
Item: _ndb_struct_na_base_pair.hbond_type_12 / _pdbx_entity_src_syn.ncbi_taxonomy_id ..._ndb_struct_na_base_pair.hbond_type_12 / _pdbx_entity_src_syn.ncbi_taxonomy_id / _pdbx_entity_src_syn.organism_scientific / _pdbx_entity_src_syn.pdbx_beg_seq_num / _pdbx_entity_src_syn.pdbx_end_seq_num |
|---|
| 改定 1.4 | 2024年5月1日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2005
ジャーナル: Nucleic Acids Res. / 年: 2005 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2hem.cif.gz
2hem.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2hem.ent.gz
pdb2hem.ent.gz PDB形式
PDB形式 2hem.json.gz
2hem.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2hem_validation.pdf.gz
2hem_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2hem_full_validation.pdf.gz
2hem_full_validation.pdf.gz 2hem_validation.xml.gz
2hem_validation.xml.gz 2hem_validation.cif.gz
2hem_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/he/2hem
https://data.pdbj.org/pub/pdb/validation_reports/he/2hem ftp://data.pdbj.org/pub/pdb/validation_reports/he/2hem
ftp://data.pdbj.org/pub/pdb/validation_reports/he/2hem リンク
リンク 集合体
集合体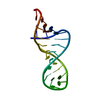
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





























 HSQC
HSQC