+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2h5d | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
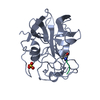
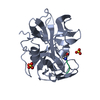
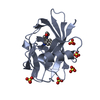
| タイトル | 0.9A resolution crystal structure of alpha-lytic protease complexed with a transition state analogue, MeOSuc-Ala-Ala-Pro-Val boronic acid | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / A-LYTIC PROTEASE / SERINE PROTEASE / ACYLATION TRANSITION STATE / CATALYSIS / PROTEIN FOLDING / PROTEIN STABILITY / PACKING DISTORTION / HYDROLASE-HYDROLASE INHIBITOR COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha-lytic endopeptidase / serine-type endopeptidase activity / proteolysis / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lysobacter enzymogenes (バクテリア) Lysobacter enzymogenes (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / REFINEMENT OF PREVIOUSLY-SOLVED STRUCTURE OF ALPHA-LYTIC PROTEASE BOUND TO MEOSUC-ALA-ALA-PRO-ALA BORONIC ACID / 解像度: 0.9 Å シンクロトロン / REFINEMENT OF PREVIOUSLY-SOLVED STRUCTURE OF ALPHA-LYTIC PROTEASE BOUND TO MEOSUC-ALA-ALA-PRO-ALA BORONIC ACID / 解像度: 0.9 Å | |||||||||
 データ登録者 データ登録者 | Fuhrmann, C.N. / Agard, D.A. | |||||||||
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2006 ジャーナル: J.Am.Chem.Soc. / 年: 2006タイトル: Subangstrom crystallography reveals that short ionic hydrogen bonds, and not a His-Asp low-barrier hydrogen bond, stabilize the transition state in serine protease catalysis 著者: Fuhrmann, C.N. / Daugherty, M.D. / Agard, D.A. #1:  ジャーナル: J.Mol.Biol. / 年: 2004 ジャーナル: J.Mol.Biol. / 年: 2004タイトル: The 0.83A resolution crystal structure of alpha-lytic protease reveals the detailed structure of the active site and identifies a source of conformational strain 著者: Fuhrmann, C.N. / Kelch, B.A. / Ota, N. / Agard, D.A. #2:  ジャーナル: Biochemistry / 年: 1989 ジャーナル: Biochemistry / 年: 1989タイトル: Structural analysis of specificity: alpha-lytic protease complexes with analogues of reaction intermediates 著者: Bone, R. / Frank, D. / Kettner, C.A. / Agard, D.A. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2h5d.cif.gz 2h5d.cif.gz | 153.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2h5d.ent.gz pdb2h5d.ent.gz | 122.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2h5d.json.gz 2h5d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2h5d_validation.pdf.gz 2h5d_validation.pdf.gz | 455.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2h5d_full_validation.pdf.gz 2h5d_full_validation.pdf.gz | 459.3 KB | 表示 | |
| XML形式データ |  2h5d_validation.xml.gz 2h5d_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  2h5d_validation.cif.gz 2h5d_validation.cif.gz | 26.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h5/2h5d https://data.pdbj.org/pub/pdb/validation_reports/h5/2h5d ftp://data.pdbj.org/pub/pdb/validation_reports/h5/2h5d ftp://data.pdbj.org/pub/pdb/validation_reports/h5/2h5d | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 19875.131 Da / 分子数: 1 / 断片: MATURE PROTEASE DOMAIN (RESIDUES 200-397) / 由来タイプ: 天然 / 詳細: gene alpha-LP / 由来: (天然)  Lysobacter enzymogenes (バクテリア) / Secretion: SECRETED BY THE NATIVE BACTERIUM / 参照: UniProt: P00778, alpha-lytic endopeptidase Lysobacter enzymogenes (バクテリア) / Secretion: SECRETED BY THE NATIVE BACTERIUM / 参照: UniProt: P00778, alpha-lytic endopeptidase | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #2: タンパク質・ペプチド | | ||||||||
| #3: 化合物 | ChemComp-SO4 / #4: 化合物 | ChemComp-GOL / | #5: 水 | ChemComp-HOH / | Has protein modification | Y | 非ポリマーの詳細 | GLYCEROL BOUND TO THE CATALYTIC ADDUCT, CREATING A MIMIC OF THE ACYLATION TRANSITION | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.44 Å3/Da / 溶媒含有率: 49.6 % |
|---|---|
| 結晶化 | 温度: 298 K / pH: 8 詳細: 1.3M LITHIUM SULFATE, 0.02M TRIS, PH 8.0, VAPOR DIFFUSION, HANGING DROP, TEMPERATURE 298K, pH 8.00 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 8.2.2 / 波長: 0.75 / ビームライン: 8.2.2 / 波長: 0.75 |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2004年6月19日 |
| 放射 | モノクロメーター: DOUBLE CRYSTAL, SI(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.75 Å / 相対比: 1 |
| 反射 | 解像度: 0.9→28.5 Å / Num. obs: 147100 / % possible obs: 99.9 % / 冗長度: 8.7 % / Rmerge(I) obs: 0.085 / Net I/σ(I): 27.9 |
| 反射 シェル | 解像度: 0.9→0.91 Å / 冗長度: 5.9 % / Rmerge(I) obs: 0.448 / Mean I/σ(I) obs: 4.8 / % possible all: 99.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: REFINEMENT OF PREVIOUSLY-SOLVED STRUCTURE OF ALPHA-LYTIC PROTEASE BOUND TO MEOSUC-ALA-ALA-PRO-ALA BORONIC ACID 開始モデル: SAME PROTEIN BOUND TO MEOSUC-ALA-ALA-PRO-ALA BORONIC ACID AT 0.9A RESOLUTION (UNPUBLISHED DATA, BUT RELATED TO 1P02) 解像度: 0.9→20 Å / Num. parameters: 20128 / Num. restraintsaints: 23688 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH & HUBER 詳細: HYDROGEN ATOMS WERE INCLUDED IN THE REFINEMENT AS "RIDING HYDROGENS", WITH POSITION AND GEOMETRY FIXED TO THOSE VALUES DEFINED BY SHELXL-97. METHYL AND HYDROXYL HYDROGENS ON SINGLE-CONFORMER ...詳細: HYDROGEN ATOMS WERE INCLUDED IN THE REFINEMENT AS "RIDING HYDROGENS", WITH POSITION AND GEOMETRY FIXED TO THOSE VALUES DEFINED BY SHELXL-97. METHYL AND HYDROXYL HYDROGENS ON SINGLE-CONFORMER SIDECHAINS WERE EACH POSITIONED AT A TORSION ANGLE THAT BEST SATISFIED POSITIVE DIFFERENCE ELECTRON DENSITY (USING INSTRUCTIONS HFIX 137 AND HFIX 147, RESPECTIVELY). IT SHOULD BE NOTED THAT THE LENGTH OF DONOR-HYDROGEN BONDS IN THIS STRUCTURE ARE LIKELY SHORTER THAN THEIR TRUE INTERNUCLEAR DISTANCE; THESE BOND LENGTHS ARE DEFINED BY SHELXL-97 PARAMETERS. THE POSITIONS OF SEVEN HYDROGEN ATOMS WERE ALLOWED TO REFINE FREELY: HIS57 HD1, HIS57 HE1, HIS57 HE2, SER214 HG, SER195 HN, GLY193 HN, AND B2V203 H1. DURING THE FINAL STAGES OF REFINEMENT, GEOMETRICAL RESTRAINTS WERE RELEASED FOR ALL NON- HYDROGEN ATOMS IN RESIDUES WITH SINGLE CONFORMATIONS. THE BORON IN RESIDUE B2V WAS REFINED AS A CARBON ATOM, ALLOWING REFINEMENT OF THE OCCUPANCY OF THIS ATOM TO ESTIMATE THE ELECTRON CONTENT (AND NEGATIVE CHARGE) AT THIS LOCATION. TO AID THE READER IN ANALYZING THE STRUCTURE IN THIS PDB FILE, THE ATOM HAS BEEN RE-NAMED TO "B", AND THE OCCUPANCY OF THE BORON ATOM MANUALLY CHANGED TO THE CORRESPONDING OCCUPANCY (1.07; REFINEMENT OF A CARBON IN THIS POSITION RESULTED IN AN OCCUPANCY OF 0.89). SEE PUBLICATION FOR MORE DETAILS.
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 21 / Occupancy sum hydrogen: 1367.28 / Occupancy sum non hydrogen: 1784.32 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 0.9→20 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj