+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2era | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | RECOMBINANT ERABUTOXIN A, S8G MUTANT | ||||||
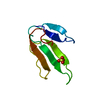
 要素 要素 | ERABUTOXIN A | ||||||
 キーワード キーワード | NEUROTOXIN / SNAKE NEUROTOXIN / VENOM / POSTSYNAPTIC NEUROTOXIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報acetylcholine receptor inhibitor activity / ion channel regulator activity / toxin activity / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  Laticauda semifasciata (エラブウミヘビ) Laticauda semifasciata (エラブウミヘビ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.81 Å 分子置換 / 解像度: 1.81 Å | ||||||
 データ登録者 データ登録者 | Gaucher, J.F. / Menez, R. / Arnoux, B. / Menez, A. / Ducruix, A. | ||||||
 引用 引用 |  ジャーナル: Eur.J.Biochem. / 年: 2000 ジャーナル: Eur.J.Biochem. / 年: 2000タイトル: High resolution x-ray analysis of two mutants of a curaremimetic snake toxin 著者: Gaucher, J.F. / Menez, R. / Arnoux, B. / Menez, A. / Ducruix, A. #1:  ジャーナル: J.Biol.Chem. / 年: 1995 ジャーナル: J.Biol.Chem. / 年: 1995タイトル: Genetic Engineering of Snake Toxins. The Functional Site of Erabutoxin A, as Delineated by Site-Directed Mutagenesis, Includes Variant Residues 著者: Tremeau, O. / Lemaire, C. / Drevet, P. / Pinkasfeld, S. / Ducancel, F. / Boulain, J.C. / Menez, A. #2:  ジャーナル: FEBS Lett. / 年: 1994 ジャーナル: FEBS Lett. / 年: 1994タイトル: Three-Dimensional Crystal Structure of Recombinant Erabutoxin a at 2.0 A Resolution 著者: Arnoux, B. / Menez, R. / Drevet, P. / Boulain, J.C. / Ducruix, A. / Menez, A. #3:  ジャーナル: J.Biol.Chem. / 年: 1993 ジャーナル: J.Biol.Chem. / 年: 1993タイトル: Genetic Engineering of Snake Toxins. Role of Invariant Residues in the Structural and Functional Properties of a Curaremimetic Toxin, as Probed by Site-Directed Mutagenesis 著者: Pillet, L. / Tremeau, O. / Ducancel, F. / Drevet, P. / Zinn-Justin, S. / Pinkasfeld, S. / Boulain, J.C. / Menez, A. #4:  ジャーナル: J.Biol.Chem. / 年: 1989 ジャーナル: J.Biol.Chem. / 年: 1989タイトル: The Crystal Structure of Erabutoxin a at 2.0-A Resolution 著者: Corfield, P.W. / Lee, T.J. / Low, B.W. #5:  ジャーナル: Acta Crystallogr.,Sect.A / 年: 1988 ジャーナル: Acta Crystallogr.,Sect.A / 年: 1988タイトル: Refinement at 1.4 A Resolution of a Model of Erabutoxin B: Treatment of Ordered Solvent and Discrete Disorder 著者: Smith, J.L. / Corfield, P.W.R. / Hendrickson, W.A. / Low, B.W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2era.cif.gz 2era.cif.gz | 25.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2era.ent.gz pdb2era.ent.gz | 15.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2era.json.gz 2era.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2era_validation.pdf.gz 2era_validation.pdf.gz | 345.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2era_full_validation.pdf.gz 2era_full_validation.pdf.gz | 345.5 KB | 表示 | |
| XML形式データ |  2era_validation.xml.gz 2era_validation.xml.gz | 2.3 KB | 表示 | |
| CIF形式データ |  2era_validation.cif.gz 2era_validation.cif.gz | 3.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/er/2era https://data.pdbj.org/pub/pdb/validation_reports/er/2era ftp://data.pdbj.org/pub/pdb/validation_reports/er/2era ftp://data.pdbj.org/pub/pdb/validation_reports/er/2era | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6823.689 Da / 分子数: 1 / 変異: S8G / 由来タイプ: 組換発現 由来: (組換発現)  Laticauda semifasciata (エラブウミヘビ) Laticauda semifasciata (エラブウミヘビ)Variant: TYPE A (N26 AND K51) / プラスミド: PRIT5 / 発現宿主:  |
|---|---|
| #2: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.8 Å3/Da / 溶媒含有率: 35 % | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.5 詳細: CRYSTALLIZATION WERE PERFORMED AT 291K BY THE HANGING DROP METHOD. DROPS OF 2 MICROLITRE OF 0.007M PROTEIN AND 2 MICROLITRE OF RESERVOIR WERE EQUILIBRATED AGAINST 3M NACL, 0.05M NAOAC BUFFER ...詳細: CRYSTALLIZATION WERE PERFORMED AT 291K BY THE HANGING DROP METHOD. DROPS OF 2 MICROLITRE OF 0.007M PROTEIN AND 2 MICROLITRE OF RESERVOIR WERE EQUILIBRATED AGAINST 3M NACL, 0.05M NAOAC BUFFER SOLUTION (PH 4.5)., vapor diffusion - hanging drop | ||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 18 ℃ / 手法: 蒸気拡散法詳細: drop solution was mixed with an equal volume of reservoir solution | ||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 278 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: LURE シンクロトロン / サイト: LURE  / ビームライン: DW32 / 波長: 0.901 / ビームライン: DW32 / 波長: 0.901 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1993年9月1日 / 詳細: MIRRORS |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.901 Å / 相対比: 1 |
| 反射 | 解像度: 1.81→15 Å / Num. obs: 4889 / % possible obs: 99.8 % / Observed criterion σ(I): 3 / 冗長度: 8.4 % / Biso Wilson estimate: 10.8 Å2 / Rsym value: 0.047 / Net I/σ(I): 13 |
| 反射 シェル | 解像度: 1.814→1.86 Å / 冗長度: 5.8 % / Mean I/σ(I) obs: 6.3 / Rsym value: 0.116 / % possible all: 97.6 |
| 反射 | *PLUS 最低解像度: 17 Å / Num. obs: 4924 / % possible obs: 99 % / Num. measured all: 41373 / Rmerge(I) obs: 0.047 |
| 反射 シェル | *PLUS % possible obs: 97 % / Rmerge(I) obs: 0.116 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: STRUCTURE OF RECOMBINANT ERABUTOXIN A PROVIDED BY DR.B.ARNOUX 解像度: 1.81→10 Å / Rfactor Rfree error: 0.01 / Data cutoff high absF: 100000 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 2 詳細: AT THE END OF REFINEMENT THE WHOLE SET OF DATA WAS USED FOR THE LAST STEPS OF REFINEMENT.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 12.1 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.18 Å / Luzzati d res low obs: 10 Å / Luzzati sigma a obs: 0.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.81→10 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.81→1.88 Å / Rfactor Rfree error: 0.032 / Total num. of bins used: 8
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / バージョン: 3.1 / 分類: refinement X-PLOR / バージョン: 3.1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS Rfactor obs: 0.193 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


