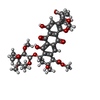
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 245d | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DNA-DRUG REFINEMENT: A COMPARISON OF THE PROGRAMS NUCLSQ, PROLSQ, SHELXL93 AND X-PLOR, USING THE LOW TEMPERATURE D(TGATCA)-NOGALAMYCIN STRUCTURE | ||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / RIGHT HANDED DNA / DOUBLE HELIX / COMPLEXED WITH DRUG | 機能・相同性 | NOGALAMYCIN / DNA |  機能・相同性情報 機能・相同性情報手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.4 Å シンクロトロン / 解像度: 1.4 Å  データ登録者 データ登録者Schuerman, G.S. / Smith, C.K. / Turkenburg, J.P. / Dettmar, A.N. / Van Meervelt, L. / Moore, M.H. |  引用 引用 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1996タイトル: DNA-drug refinement: a comparison of the programs NUCLSQ, PROLSQ, SHELXL93 and X-PLOR, using the low-temperature d(TGATCA)-nogalamycin structure. 著者: Schuerman, G.S. / Smith, C.K. / Turkenburg, J.P. / Dettmar, A.N. / Van Meervelt, L. / Moore, M.H. #1:  ジャーナル: Biochemistry / 年: 1995 ジャーナル: Biochemistry / 年: 1995タイトル: DNA-Nogalamycin Interactions: The Crystal Structure of d(TGATCA) Complexed with Nogalamycin 著者: Smith, C.K. / Davies, G.J. / Dodson, E.J. / Moore, M.H. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  245d.cif.gz 245d.cif.gz | 32.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb245d.ent.gz pdb245d.ent.gz | 22.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  245d.json.gz 245d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  245d_validation.pdf.gz 245d_validation.pdf.gz | 553.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  245d_full_validation.pdf.gz 245d_full_validation.pdf.gz | 568.6 KB | 表示 | |
| XML形式データ |  245d_validation.xml.gz 245d_validation.xml.gz | 3.7 KB | 表示 | |
| CIF形式データ |  245d_validation.cif.gz 245d_validation.cif.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/45/245d https://data.pdbj.org/pub/pdb/validation_reports/45/245d ftp://data.pdbj.org/pub/pdb/validation_reports/45/245d ftp://data.pdbj.org/pub/pdb/validation_reports/45/245d | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 1808.229 Da / 分子数: 2 / 由来タイプ: 合成 #2: 化合物 | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.42 Å3/Da / 溶媒含有率: 64.02 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.5 / 詳細: pH 6.50, VAPOR DIFFUSION, HANGING DROP | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 6.5 / 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 150 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SRS SRS  / ビームライン: PX9.6 / 波長: 0.87 / ビームライン: PX9.6 / 波長: 0.87 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE |
| 放射 | 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.87 Å / 相対比: 1 |
| 反射 | 最高解像度: 1.4 Å / Num. all: 62880 / Num. obs: 10306 / Observed criterion σ(I): 0 / Rmerge(I) obs: 0.036 |
| 反射 | *PLUS Num. obs: 2230 / % possible obs: 97.5 % / Num. measured all: 13447 / Rmerge(I) obs: 0.056 |
| 反射 シェル | *PLUS 最高解像度: 2.8 Å / 最低解像度: 2.9 Å / % possible obs: 86.8 % / Rmerge(I) obs: 0.21 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.4→8 Å / σ(F): 0 詳細: DISTANCES AND ANGLES WITHIN THE BASES WERE RESTRAINED TO TARGET VALUES. ALL OTHER CHEMICALLY EQUIVALENT BOND DISTANCES WERE RESTRAINED TO BE SIMILAR BY SAME DISTANCE RESTRAINTS. SIGMAS FOR ...詳細: DISTANCES AND ANGLES WITHIN THE BASES WERE RESTRAINED TO TARGET VALUES. ALL OTHER CHEMICALLY EQUIVALENT BOND DISTANCES WERE RESTRAINED TO BE SIMILAR BY SAME DISTANCE RESTRAINTS. SIGMAS FOR BOTH TARGET AND SIMILAR DISTANCE RESTRAINTS WERE SET AT 0.030 ANGSTROMS FOR BONDS AND 0.050 ANGSTROMS FOR ANGLES. SADI_* 0.15 C3'_- P C3'_1 P_2 SADI_* 0.15 C5' P C5'_2 P_2 ISOR 0.1 ISOR 0.05 C2_3 C2_5 ISOR 0.025 C15B_41 DELU C15B_41 O10B_41 C14_41 C10_41 C20_ 41 DELU O5'_21 C5'_21 C4'_21 C3'_21 C2'_21 C1'_21 O4'_21 BUMP
| |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 18.8 Å2 | |||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.2 Å / Luzzati sigma a obs: 0.17 Å | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.4→8 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-93 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: s_angle_deg / Dev ideal: 4.22 |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj