+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 225d | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
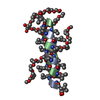
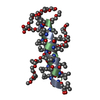
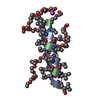
| タイトル | A TETRAMERIC DNA STRUCTURE WITH PROTONATED CYTOSINE:CYTOSINE BASE PAIRS | ||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / INTERCALATED CYTOSINE TETRAD | 機能・相同性 | DNA |  機能・相同性情報 機能・相同性情報手法 | 溶液NMR / MOLECULAR DYNAMICS, ENERGY MINIMIZATION |  データ登録者 データ登録者Gehring, K. / Leroy, J.L. / Gueron, M. |  引用 引用 ジャーナル: Nature / 年: 1993 ジャーナル: Nature / 年: 1993タイトル: A tetrameric DNA structure with protonated cytosine.cytosine base pairs. 著者: Gehring, K. / Leroy, J.L. / Gueron, M. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  225d.cif.gz 225d.cif.gz | 21.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb225d.ent.gz pdb225d.ent.gz | 14.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  225d.json.gz 225d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  225d_validation.pdf.gz 225d_validation.pdf.gz | 237 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  225d_full_validation.pdf.gz 225d_full_validation.pdf.gz | 236.8 KB | 表示 | |
| XML形式データ |  225d_validation.xml.gz 225d_validation.xml.gz | 1.7 KB | 表示 | |
| CIF形式データ |  225d_validation.cif.gz 225d_validation.cif.gz | 2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/25/225d https://data.pdbj.org/pub/pdb/validation_reports/25/225d ftp://data.pdbj.org/pub/pdb/validation_reports/25/225d ftp://data.pdbj.org/pub/pdb/validation_reports/25/225d | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 1705.145 Da / 分子数: 4 / 由来タイプ: 合成 / 詳細: CHEMICALLY SYNTHESIZED /  キーワード: DEOXYRIBONUCLEIC ACID キーワード: DEOXYRIBONUCLEIC ACID |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: DATA COLLECTION PARAMETERS: FREQUENCY: 600 MHZ FOR 1H, 242 MHZ FOR 31P SAMPLE TUBE: 5 MM, 7.5 MILLIMOLAR STRAND, PH 4.3, 25 C SPECTRA FOR ASSIGNMENTS: HOMONUCLEAR PROTON 2D, 1H-31P TOCSY; ...Text: DATA COLLECTION PARAMETERS: FREQUENCY: 600 MHZ FOR 1H, 242 MHZ FOR 31P SAMPLE TUBE: 5 MM, 7.5 MILLIMOLAR STRAND, PH 4.3, 25 C SPECTRA FOR ASSIGNMENTS: HOMONUCLEAR PROTON 2D, 1H-31P TOCSY; SPECTRA FOR CONSTRAINTS: NOESY, DQF-COSY, 1H-31P COSY |
- 試料調製
試料調製
| 試料状態 | pH: 4.3 / 温度: 298 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
- 解析
解析
| ソフトウェア |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| NMR software | 名称:  X-PLOR / 開発者: BRUNGER / 分類: 精密化 X-PLOR / 開発者: BRUNGER / 分類: 精密化 | ||||||||
| 精密化 | 手法: MOLECULAR DYNAMICS, ENERGY MINIMIZATION / ソフトェア番号: 1 詳細: STRUCTURE CALCULATION: MOLECULAR DYNAMICS AND ENERGY MINIMIZATION USING X-PLOR CONSTRAINTS (PER STRAND): TOTAL 70 DISTANCE CONSTRAINTS (ABOUT 12/NUCLEOTIDE) 43 FROM INTRANUCLEOTIDE NOES 6 ...詳細: STRUCTURE CALCULATION: MOLECULAR DYNAMICS AND ENERGY MINIMIZATION USING X-PLOR CONSTRAINTS (PER STRAND): TOTAL 70 DISTANCE CONSTRAINTS (ABOUT 12/NUCLEOTIDE) 43 FROM INTRANUCLEOTIDE NOES 6 FROM SEQUENTIAL INTERNUCLEOTIDE NOES 21 FROM NON-SEQUENTIAL INTERNUCLEOTIDE NOES 15 FROM HYDROGEN BONDS DIHEDRAL ANGLE CONSTRAINTS 10 BACKBONE AND SUGAR RING TORSION ANGLES CONSTRAINTS TO MAKE CYTOSINE BASE PAIRS PLANAR 4-FOLD SYMMETRY CONSTRAINTS (AS 3 SETS OF PAIRWISE NON-CRYSTALLOGRAPHIC SYMMETRY CONSTRAINTS) | ||||||||
| NMR constraints | NOE constraints total: 70 / NOE intraresidue total count: 43 / NOE sequential total count: 6 / Hydrogen bond constraints total count: 15 | ||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj

